This is an old revision of the document!
Ateliers R du CSBQ
Cette série de 10 ateliers guide les participants à travers les étapes requises afin de maîtriser le logiciel R pour une grande variété d’analyses statistiques pertinentes en recherche en biologie et en écologie. Ces ateliers en libre accès ont été créés par des membres du CSBQ à la fois pour les membres du CSBQ et pour la grande communauté d’utilisateurs de R.
Le contenu de cet atelier a été révisé par plusieurs membres du CSBQ. Si vous souhaitez y apporter des modifications, veuillez SVP contacter les coordonnateurs actuels de la série, listés sur la page d'accueil
Atelier 9: Analyses multivariées
Développé par : Bérenger Bourgeois, Xavier Giroux-Bougard, Amanda Winegardner, Emmanuelle Chrétien et Monica Granados. (Le script R est en parti issu de: Borcard, Gillet & Legendre (2011). Numerical Ecology with R. Springer New York).
Résumé: Dans cet atelier, vous apprendrez les bases des analyses multivariées qui vous permettront de révéler les patrons de diversité dans vos données de communautés. Vous apprendrez d'abord comment choisir les mesures de distance et les transformations appropriées pour ensuite réaliser plusieurs types d'analyses multivariées: des groupements, des Analyses en Composantes Principales (PCA), des Analyses de Correspondance (CA), des Analyses en Coordonnées Principales (PCoA) et des Positionnements Multidimensionnels Non-Métriques (NMDS).
Lien vers la nouvelle présentation Rmarkdown
Lien vers la présentation Prezi associée : Prezi
Téléchargez le script R et les données pour cet atelier:
Télechargez les paquets R pour cet atelier:
- | Chargez les paquets et les fonctions nécessaires
install.packages("vegan") install.packages("gclus") install.packages("ape") library(vegan) library(gclus) library(ape) source(file.choose()) #coldiss.R
Qu'est-ce que l'ordination?
L'ordination est un ensemble de méthodes pour décrire des échantillons dans de multiples dimensions (Clarke et Warwick 2011). Les méthodes d'ordination sont donc très utiles pour simplifier et interpréter des données multivariées. Les écologistes parlent souvent de “faire une PCA” face à des données multidimensionnelles complexes et désordonnées. Programmer des méthodes d'ordination à partir de R est relativement simple. L'interprétation des analyses d'ordination peut par contre être plus difficile, surtout si vous n'êtes pas sûr des questions biologiques que vous souhaitez explorer avec la méthode d'ordination que vous utilisez. Un examen attentif des objectifs de ces méthodes et de leur cadre d'application est nécessaire pour obtenir de bons résultats !
Lorsque vous utilisez une méthode d'ordination, un ensemble de variables est utilisé pour ordonner des échantillons (objets, sites, etc.) le long d'axes principaux représentant des combinaison de variables (Gotelli et Ellison 2004). L'ordination permet donc de réduire ou de simplifier les données en créant de nouveaux axes intégrant la majeure partie de la variation présentes dans les données. A titre d'exemple, un ensemble de données avec 24 variables peut être réduit à cinq composantes principales qui représentent les principaux gradients de variation entre les échantillons. Les méthodes d'ordination sans contrainte ne sont pas adaptées au test d'hypothèses biologiques, mais permettent l'analyse exploratoire des données. Voir (the Ordination Website) pour un aperçu des différents méthodes (en Anglais).
Introduction aux données
Nous allons utiliser deux principaux ensembles de données dans la première partie de cet atelier. “DoubsSpe.csv” est une matrice de données d'abondance d'espèces de communautés de poissons dans laquelle la première colonne contient les noms des sites de 1 à 30 et les colonnes subséquentes correspondent aux différentes espèces de poissons. “DoubsEnv.csv” est une matrice de données environnementales pour les mêmes sites. La première colonne contient donc les noms des sites de 1 à 30 et les colonnes suivantes les mesures de 11 variables abiotiques. Notez que les données utilisées pour les analyses d'ordination sont généralement en format long (en anglais).
- | Chargez DoubsSpe et DoubsEnv
#Matrice d'abondances d'espèces: “DoubsSpe.csv” spe<- read.csv(file.choose(), row.names=1) spe<- spe[-8,] #Pas d'espèces dans le site 8, supprimer site 8. #Exécutez cette ligne une seule fois. #L'environnement: “DoubsEnv.csv” env<- read.csv(file.choose(), row.names=1) env<- env[-8,] #Supprimer site 8 puisqu'on l'a retiré de la matrice d'abondances. #Exécutez cette ligne une seule fois.
1. Exploration des données
1.1 Données sur les espèces
Nous pouvons utiliser les fonctions de résumé R pour explorer les données “Spe” (données d'abondances de poissons) et découvrir les caractéristiques telles que les dimensions de la matrice, les noms des colonnes et les statistiques descriptives de ces colonnes (révision de l'atelier 2).
- | Explorez DoubsSpe
names(spe) # Les noms des colonnes dim(spe) # Le nombre de lignes et de colonnes. str(spe) # La structure interne de la matrice. head(spe) # Les premières lignes. summary(spe) # Les statistiques descriptives.
Regardez la distribution des espèces.
- | La distribution des espèces (DoubsSpe)
(ab<-table(unlist(spe))) #Les parenthèses signifient que la sortie s'affiche immédiatement barplot(ab, las=1, xlab=”Abundance class”, ylab=”Frequency”, col=grey(5:0/5))
 Pouvez-vous voir qu'il y a une grande fréquence de zéros dans les données d'abondance ?
Pouvez-vous voir qu'il y a une grande fréquence de zéros dans les données d'abondance ?
Calculez le nombre d'absences.
- | Absences
sum(spe==0)
Regardez la proportion de zéros dans les données de la communauté de poissons.
- | Proportion de zéros
sum(spe==0)/(nrow(spe)*ncol(spe))
La proportion de zéros dans la matrice est de ~0.5
Calculez le nombre de sites où chaque espèce est présente.
- | Nombre de sites où chaque espèce est présente
spe.pres<- colSums(spe>0) # Somme des sites où chaque espèce est présente. hist(spe.pres, main=”Cooccurrence des espèces”, las=1, xlab=”Fréquence”, breaks=seq(0,30, by=5), col=”grey”)
Le plus grand nombre d'espèces se retrouvent dans un nombre intermédiaire de sites.
Calculez le nombre d'espèces présentes à chaque site. Ici, nous utilisons une façon simpliste de calculer la richesse en espèces. Dans certains cas, le nombre d'espèces présentes dans un site varie entre les sites car il peut y avoir une relation entre le nombre d'individus comptés (l'abondance) dans ce site et la richesse en espèces. richesse d'espèces raréfiée (en anglais) est souvent une mesure plus appropriée que la richesse totale. La fonction rarefy () de vegan peut être utilisée pour calculer la richesse raréfiée des espèces.
- | Richesse en espèces
site.pres<- rowSums(spe>0) # Nombre d'espèces présentes dans chaque site hist(site.pres, main=”Richesse en espèces”, las=1, xlab=”Fréquence des sites”, ylab=”Nombre d'espèces”, breaks=seq(0,30, by=5), col=”grey”)
1.2 Données sur l'environnement
Explorez les données environnementales pour détecter les colinéarités :
- | Exploration de DoubsEnv
names(env) dim(env) str(env) head(env) summary(env) pairs(env, main="Données environnementales" )
Dans ce cas, les données environnementales sont toutes dans des unités différentes et doivent donc être standardisées avant de calculer les mesures de distance utilisées pour effectuer la plupart des analyses d'ordination. La standardisation des données (11 variables) peut être effectuée en utilisant la fonction decostand () de vegan.
- | Standardisation des données
env.z<-decostand(env, method="standardize") apply(env.z, 2, mean) # Les données sont maintenant centrées (moyennes~0)... apply(env.z, 2, sd) # et réduites (écart-type=1)
2. Mesures d'association
L'algèbre matricielle est à la base des méthodes d'ordination. Une matrice est constituée de données (ex. valeurs mesurées) réparties en lignes et colonnes. Les analyses d'ordination sont effectuées sur des matrices d'association calculées à partir des matrices de données écologiques (telles que DoubsEnv ou DoubsSpe). La création d'une matrice d'association permet le calcul de similarité et de distance entre les objets ou les descripteurs (Legendre et Legendre 2012). Avant de se lancer dans des analyses d'ordination, il est important de passer du temps sur vos matrices de données. Explorer les mesures possibles d'association qui peuvent être générées à partir de vos données avant de faire une ordination peut vous aider à mieux comprendre quelles mesures de distance sont appropriées pour les méthodes d'ordination. Il peut être difficile de voir l'objectif de chaque indice de dissimilarité, mais cette connaissance sera nécessaire pour mieux comprendre les méthodes d'ordination canoniques présentées par la suite.
EN RÉSUMÉ: Pour l'ordination d'objets, il faut calculer les distances entre eux. Ces distances peuvent être calculées de plusieurs façons, en prenant en compte l'abondance ou les données de présence / absence. Plus important encore, plusieurs propriétés sont de grande importance pour les mesures de distances, et elles seront explorées dans les exemples ci-dessous. Pour plus d'informations sur les propriétés des mesures de distance et certains termes clés, cliquez sur la section cachée.
2.1 Mesures de distance
Les données quantitatives des espèces Nous pouvons utiliser la fonction vegdist () pour calculer des indices de dissimilarité sur des données de composition de la communauté. Ceux-ci peuvent ensuite être visualisés sous forme de matrice si désiré.
- | vegdist
spe.db<-vegdist(spe, method="bray") # distance de Bray (avec des données de présence-absence, correspond à Sorensen) spe.dj<-vegdist(spe, method="jac") # distance de Jaccard spe.dg<-vegdist(spe, method="gower") # distance de Gower spe.db<-as.matrix(spe.db) # réarranger en format matrice (pour visualisation, ou pour exporter en .csv)
Une version condensée de la matrice spe.db représentant la distance entre les trois premières espèces de DoubsSpe ressemblerait à ceci:
| Espèce 1 | Espèce 2 | Espèce 3 | |
|---|---|---|---|
| Espèce 1 | 0.0 | 0.6 | 0.68 |
| Espèce 2 | 0.6 | 0.0 | 0.14 |
| Espèce 3 | 0.68 | 0.14 | 0.0 |
Vous pouvez voir que lorsque l'on compare une espèce à elle-même (par exemple, Espèce 1 à Espèce 1), la distance est de 0 puisque les espèces sont identiques.
Ces mêmes mesures de distance peuvent être calculées à partir des données de présence-absence par l'utilisation de l'argument binary=TRUE dans la fonction vegdist(). Cela donnera des mesures de distance légèrement différentes.
Vous pouvez également créer des représentations graphiques de ces matrices d'association en utilisant la fonction coldiss.
Données environnementales quantitatives Regardons les associations entre les variables environnementales (aussi appelée mode Q):
- | distances mesurées à partir des données environnementales
env.de<-dist(env.z, method = "euclidean") # matrice de distances euclidiennes des données env. standardisées windows() # crée une nouvelle fenêtre graphique coldiss(env.de, diag=TRUE)
Nous pouvons ensuite regarder la dépendance entre les variables environnementales (aussi appelée mode R):
- | corrélations entre les données environnementales
(env.pearson<-cor(env)) # coefficient r de corrélation de Pearson round(env.pearson, 2) # arrondit les coefficients à deux décimales (env.ken<-cor(env, method="kendall")) # coefficient tau de corrélation de rang de Kendall round(env.ken, 2)
La corrélation de Pearson mesure la corrélation linéaire entre deux variables. La corrélation de Kendall est une corrélation de rang qui quantifie la relation entre deux descripteurs ou deux variables lorsque les données sont ordonnées au sein de chaque variable.
Dans certains cas, il peut y avoir des types mixtes de variables environnementales. Le mode Q peut alors être utilisé pour trouver des associations entre variables environnementales. C'est ce que nous allons faire avec l'exemple fictif suivant:
- | Exemple
var.g1<-rnorm(30, 0, 1) var.g2<-runif(30, 0, 5) var.g3<-gl(3, 10) var.g4<-gl(2, 5, 30) (dat2<-data.frame(var.g1, var.g2, var.g3, var.g4)) str(dat2) summary(dat2)
Une matrice de dissimilarité peut être générée pour ces variables mixtes en utilisant la distance de Gower:
- | daisy
?daisy #Cette fonction peut gérer la présence de NA dans les données (dat2.dg<-daisy(dat2, metric="gower")) coldiss(dat2.dg)
Défi 1 - Niveau intermédiaire Discutez avec votre voisin: Comment pouvons-nous dire si des objets sont similaires avec un jeu de données multivariées? Faites une liste de toutes vos suggestions.
Défi 1 - Solution
Défi 1 - Niveau avancé Calculer à la mitaine sans utiliser la fonction decostand () les distances de Bray-Curtis et de Gower pour l'abondance des espèces CHA, TRU et VAI dans les sites 1, 2 et 3.
Défi 1 - Solution
2.2 Transformations des données de composition des communautés
Les données de composition des communautés peuvent également être standardisées ou transformées. La fonction decostand () de vegan fournit des options de standardisation et de transformation de ce type de données
Transformer les abondances en données de présence-absence:
- | decostand, présence-absence
spe.pa<-decostand(spe, method="pa")
D'autres transformations peuvent être utilisées pour corriger l'influence d'espèces rares, par exemple, la transformation de Hellinger:
- | Hellinger et Chi-carré
#La transformation Hellinger spe.hel<-decostand(spe, method="hellinger") # vous pouvez aussi simplement écrire "hel" #Transformation de chi-carré spe.chi<-decostand(spe, method="chi.square")
Défi option 2 - Niveau avancé Calculez les distances de Hellinger et de Chi-carré sur les données “spe” sans utiliser decostand ().
Défi option 2 - Solution
2.3 Groupement
Les matrices d’association nécessaires pour utiliser les méthodes de groupement. Le groupement n’est pas une méthode statistique en tant que telle puisqu’elle ne teste pas d’hypothèse, mais permet de déceler des structures dans les données en partitionnant soit les objets, soit les descripteurs. Les objets similaires sont agrégés en sous-groupes ce qui permet de mettre en relief des cassures (contrastes) entre les données. En tant que biologiste, il peut être intéressant de tenter de séparer une série de sites en groupes en fonction de leurs caractéristiques environnementales ou de leur composition en espèces.
Les résultats d’un groupement sont généralement représentés sous forme de dendrogramme (arbre), dans lequel les objets sont agrégés en groupes. Il y a plusieurs familles de méthodes de groupements, mais nous présenterons uniquement un aperçu de trois méthodes : le groupement agglomératif hiérarchique à liens simples, le groupement agglomératif hiérarchique à liens complets et la méthode de Ward. Pour plus de détails sur les différentes familles de méthodes de groupement, consulter Legendre et Legendre 2012 (chapitre 8).
Dans les méthodes hiérarchiques, les éléments des petits ensembles se regroupent en groupes plus vastes de rang supérieur, et ainsi de suite (par exemple : espèces, genres, familles, ordre). Avant de faire le groupement, il faut créer une matrice d’association entre les objets. Une matrice de distances est le choix par défaut des fonctions de groupement dans R. La matrice d’association est premièrement classée en ordre croissant de distances. Ensuite, les groupes sont formés de manière hiérarchique selon les critères spécifiques à chaque méthode.
Prenons un exemple tout simple d'une matrice de distances euclidiennes entre 5 objets dont on a ordonné les distances en ordre croissant.
Pour le groupement agglomératif à liens simples, les deux objets les plus proches se regroupent en premier. Ensuite, un deuxième groupe est formé à partir des deux objets les plus proches suivant (il se peut que ce soit deux objets différents, ou bien un objet et le groupe formé précédemment), et ainsi de suite. Cette méthode forme généralement de longues chaînes de groupes (dans l'exemple ci-haut, les objets 1 à 5 se regroupent successivement). À l’inverse, pour le groupement agglomératif à liens complet, un objet se regroupe à un autre objet/groupe seulement lorsqu’il est aussi lié à l’élément le plus éloigné de ce groupe. Ainsi, quand deux groupes fusionnent, tous les éléments des deux groupes sont liés à la distance considérée (ci-haut, le groupe 3-4 ne se lie au groupe 1-2 qu'à la distance à laquelle tous les autres éléments sont déjà liés). C’est pour cette raison que le groupement à liens complets forme généralement plusieurs petits groupes séparés et qu’elle peut être plus appropriée pour relever des contrastes ou des discontinuités dans les données.
Comparons ces deux méthodes en utilisant les données d’abondances de poissons de la rivière Doubs. Les données d’abondances ont été au préalable transformées par la méthode Hellinger. Puisque les fonctions de groupement requièrent une matrice de distances, la première étape sera de générer une matrice de distances Hellinger.
- | vegdist, distance de Hellinger
spe.dhel<-vegdist(spe.hel,method="euclidean") #crée une matrice de distances Hellinger à partir des données d’abondance transformées #Pour voir la différence entre les deux types d’objets head(spe.hel)# données d’abondances transformées Hellingerhead(spe.dhel)# matrice de distances de Hellinger entre les sites
La plupart des méthodes de groupement sont disponible dans la fonction hclust() de la librairie stats
- | hclust, comparaison du groupement à liens simples et à liens complets
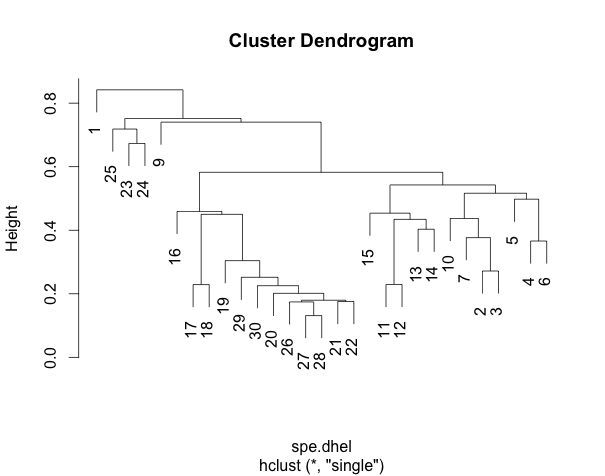
#Faire le groupement à liens simples spe.dhel.single<-hclust(spe.dhel, method="single") plot(spe.dhel.single) #Faire le groupement à liens complet spe.dhel.complete<-hclust(spe.dhel, method="complete") plot(spe.dhel.complete)
Est-ce que les deux dendrogrammes sont très différents?
On remarque que pour le groupement à liens simple, plusieurs objets s’enchaînent (par exemple les sites 19, 29, 30, 20, 26, etc.) alors que des groupes plus distincts peuvent être observés dans le groupement à liens complets.
La méthode de Ward diffère légèrement des deux méthodes précédentes. Le critère utilisé est la méthode des moindres carrés (comme dans les modèles linéaires). Ainsi, des objets/groupes fusionnent de façon à minimise la variance intragroupes. Pour débuter, chaque objet est considéré comme un groupe. À chaque étape, la paire de groupes à fusionner est celle qui résulte à la plus petite augmentation de la somme des carrés des écarts intra-groupes.
La méthode de Ward est également disponible sous la fonction hclust(). Par contre, le dendrogramme produit par défaut montre les distances au carré. Afin de comparer ce dendrogramme à celui du groupement à liens simples et à liens complets, il faut calculer la racine carrée des distances.
- | hclust, méthode de Ward
#Faire le groupement de Ward spe.dhel.ward<-hclust(spe.dhel, method="ward.D2") plot(spe.dhel.ward) #Refaire le dendrogramme en utilisant la racine carrée des distances spe.dhel.ward$height<-sqrt(spe.dhel.ward$height) plot(spe.dhel.ward) plot(spe.dhel.ward, hang=-1) # hang=-1 permet d’afficher les objets sur la même ligne
Les groupements générés par la méthode de Ward ont tendance à être plus sphériques et à contenir des quantités plus similaires d’objets.
Quelle méthode choisir?
Le choix de la bonne mesure d’association et de la bonne méthode de groupement dépend de l’objectif. Qu’est-ce qu’il est plus intéressant de démontrer : des gradients? des contrastes? Il est également important de tenir en compte les propriétés de la méthode utilisée dans l’interprétation des résultats. Si plus d’une méthode semble adéquate pour répondre à une question biologique, comparer les dendrogrammes serait une bonne option. Encore une fois, le groupement n’est pas une analyse statistique, mais il est possible de tester les résultats et d’identifier des partitions ayant un sens biologique. Il est également possible de déterminer le nombre de groupes optimal et de performer des tests statistiques sur les résultats. Les méthodes de groupement peuvent aussi être combinées à une ordination pour distinguer des groupes de sites. Ces avenues ne seront pas explorées dans cet atelier. Pour aller plus loin, consulter Borcard et al. 2011.
3. Ordination sans contrainte
Les analyses d'ordination sans contrainte permettent d'organiser des échantillons, des sites ou des espèces le long de gradients continus (ex. écologiques ou environnementaux). Les ordinations sans contrainte se différencient des analyses canoniques (voir plus loin dans cet atelier “analyses canoniques”) par le fait que ces techniques ne tentent pas de définir une relation entre des variables dépendantes et indépendantes.
L'ordination sans contrainte peut être utilisée pour: - Évaluer les relations au sein d'un ensemble de variables (et non pas entre séries de variables). - Trouver les éléments clés de variation au sein d'échantillons, de sites, d'espèces, etc. - Réduire les dimensions d'un jeu de données multivariées sans perte importante d'informations. - Créer de nouvelles variables pour une utilisation dans des analyses ultérieures (comme la régression). Les composantes principales des axes d'ordination sont en effet des combinaisons linéaires des variables d'origine. Source
3.1 Analyses en Composantes Principales (Principal Component Analysis, PCA)
L'Analyse en Composantes Principales (ou PCA) fur originellement décrite par Pearson (1901) bien qu'elle soit le plus souvent attribué à Hotelling (1933) qui l'a proposé indépendamment. Cette méthode ainsi que nombre de ces implications sont pour l'analyse de données sont présentées dans l'article fondateur de Rao (1964). La PCA est utilisée pour générer, à partir d'un large jeu de données, un nombre restreint de variables clefs qui permettent de représenter au maximum la variation présente dans le jeu de données. En d'autres termes, la PCA est utilisée pour générer des combinaisons de variables à partir d'un ensemble plus grand de variables tout en conservant la majorité de variation de l'ensemble des données. La PCA est une technique d'analyse puissante pour l'analyse dans descripteurs quantitatifs (tels que les abondances d'espèces), mais ne peut pas être appliquée aux données binaires (telles que l'absence/présence des espèces).
À partir d'un jeu de données contenant des variables à distribution normale, le premier axe de PCA (ou axe de composante principale) correspond à la droite qui traverse la plus grande dimension de l’ellipsoïde décrivant la distribution multi-normale des données. Les axes suivants traversent de façon similaire cet ellipsoïde selon l'ordre décroissant de ces dimensions. Ainsi, il est possible d'obtenir un maximum de p axes principaux à partir d'un jeu de données contenant p variables.
Pour cela, la PCA effectue un rotation du système d'axes originels défini par les variables de façon à ce que les axes d'ordination successifs soient orthogonaux entre eux et correspondent aux dimensions successives du maximum de variance observée dans le nuage de points (voir ci-dessus). Les nouvelles variables produites par la PCA sont non-corrélées entre elles (les axes d'ordination étant orthogonaux) et peuvent alors être utilisés dans d'autres types d'analyse telles que des régressions multiples (Gotelli et Ellison, 2004). Les composantes principales situent la position des objets dans le nouveau système de coordonnées calculé par la PCA. La PCA s'effectue sur une matrice d'association entre variables, et a pour caractéristique de préserver les distances euclidiennes et de détecter des relations linéaires, uniquement. En conséquence, les abondances brutes des espèces doivent être soumises à une pré-transformation (comme une transformation d'Hellinger) avant de réaliser une PCA.
Pour faire une PCA, vous avez besoin : - Un ensemble de variables (sans distinction entre les variables indépendantes ou dépendantes, c-est-à-dire un ensemble d'espèces OU un ensemble de variables environnementales). - De sites (objets, échantillons) dans lesquels sont mesurés les mêmes variables. - En général, il est préférable d'avoir un plus grand nombre de sites que de variables dans le jeu de données (plus de lignes que de colonnes).
La PCA est particulièrement utile pour des matrices de données contenant plus de deux variables, mais il est plus facile de décrire son fonctionnement avec un exemple bidimensionnel. Dans l'exemple suivant (d'après Clarke et Warwick 2001), la matrice contient les données d'abondance de deux espèces dans neuf sites:
| Site | Espèce 1 | Espèce 2 |
|---|---|---|
| A | 6 | 2 |
| B | 0 | 0 |
| C | 5 | 8 |
| D | 7 | 6 |
| E | 11 | 6 |
| F | 10 | 10 |
| G | 15 | 8 |
| H | 18 | 14 |
| I | 14 | 14 |
La représentation des sites en deux dimensions devrait ressembler à ceci:

Ce nuage de points est une ordination. Il présente la distribution des espèces entre les sites, mais vous pouvez imaginer qu'il est plus difficile de visualiser un tel graphique en présence de plus de deux espèces. Dans ce cas, l'objectif est de réduire le nombre de variables en composantes principales. Pour réduire les données bidimensionnelles précédentes à une dimension, une PCA peut être effectuée :
Dans ce cas, la première composante principale est orientée dans le sens de la plus grande variation dans les points, ces points étant perpendiculaires à la ligne.
Une seconde composante principale est alors ajoutée perpendiculairement à la première:
Dans le diagramme final, les deux axes de PCA sont pivotés et les axes sont maintenant les composantes principales (et non plus les espèces):
Pour les PCA avec plus de deux variables, les composantes principales sont ajoutées de la façon suivante (Clarke et Warwick 2001):
PC1 = axe qui maximise la variance des points qui sont projetées perpendiculairement à l'axe. PC2 = axe perpendiculaire à PC1, mais dont la direction est à nouveau celle maximisant la variance lorsque les points y sont projetés perpendiculairement. PC3 et ainsi de suite: perpendiculaire aux deux premiers axes dont la direction est à nouveau celle maximisant la variance lorsque les points y sont projetés perpendiculairement.
Lorsqu'il y a plus de deux dimensions, la PCA produit un nouvel espace dans lequel tous les axes de PCA sont orthogonaux (ce qui signifie que la corrélation entre chaque combinaison de deux axes est nulle), et où les axes de PCA sont ordonnés selon la proportion de variance des données d'origine qu'ils expliquent.
Les données “spe” comprennent 27 espèces de poissons. Pour simplifier cette diversité à un petit nombre de variables ou pour identifier différents groupes de sites associés à des espèces particulières, une PCA peut être effectuée.
Exécuter une PCA sur les données d'abondance d'espèces soumises à la transformation d'Hellinger :
- | PCA avec rda() de (vegan)
#Exécuter la PCA avec la fonction rda()- cette fonction calcule à la fois des PCA et des RDA spe.h.pca<-rda(spe.hel) #Extraire les résultats summary(spe.h.pca)
Résultats:
Interprétation des résultats de PCA Cette sortie R contient les valeurs propres ou «eigenvalue» de la PCA. La valeur propre est la valeur de la variation ramenée à la longueur d'un vecteur, et correspond à la quantité de variation expliquée par chaque axe d'ordination de la PCA. Comme vous pouvez le voir, la fonction summary fournit de nombreuses informations. Parmi les résultats, la proportion de variance des données expliquée par les variables sans contraintes est une information importante. Dans cet exemple, la variance totale des sites expliquée par les espèces est de 0,5 (50%). Le résumé vous indique également quelle proportion de la variance totale expliquée est répartie entre chaque composantes principales de la PCA: le premier axe de PCA explique 51.33% de la variation tandis que le second axe explique 12.78%. Vous pouvez également extraire certaines parties des résultats:
- | Résultats de PCA
summary(spe.h.pca, display=NULL) # seulement les valeurs propres eigen(cov(spe.hel)) # vous pouvez aussi trouver les valeurs propres par cette ligne de code
Les scores (c'est-à-dire les coordonnées) des sites ou des espèces peuvent également être extraits d'une PCA. Ces scores permettent, par exemple, d'utiliser une composante principale comme une variable dans une autre analyse, ou de faire des graphiques supplémentaires. Par exemple, vous pouvez obtenir par PCA une variable unique issue du jeu de données “spe” puis l'utiliser pour la corréler par régression à une autre variable, ou déterminer un gradient spatial. Pour extraire les scores d'une PCA, utiliser la fonction scores ():
- | scores()
spe.scores<-scores(spe.h.pca, display="species", choices=c(1,2)) # scores des espèces selon les premier et deuxième axes site.scores<-scores(spe.h.pca, display="sites", choices=c(1,2)) # scores des sites selon les premier et deuxième axes #Remarque: si vous ne spécifiez pas le nombre de composantes principales à l'aide de choices = c (1,2) #(ou choices = c (1: 2)), les scores selon toutes les composantes principales seront extraits.
La PCA des données d'abondances de poissons produit autant de composantes principales qu'il y d'espèces (i.e. de colonnes dans le jeu de données), soit 27 composantes principales. Le nombre de variables à traiter n'est donc pas directement réduit par la PCA. Pour réduire le nombre de variables, il est alors nécessaire de déterminer quelles composantes principales sont significatives et doivent être conservées, par exemple à l'aide du critère de Kaiser-Guttman. Ce critère compare la variance expliquée par chaque composante principale à la moyenne de la variance expliquée par l'ensemble des composantes principales. Un histogramme illustrant la significativité des différentes composantes principale peut ensuite être tracé à l'aide du code ci-dessous :
- | Les axes
# Identification des axes significatifs de la PCA à l'aide du critère de Kaiser-Guttman ev<-spe.h.pca$CA$eig ev[ev>mean(ev)] n<-length(ev) bsm<-data.frame(j=seq(1:n), p=0) bsm$p[1]=1/n for (i in 2:n) { bsm$p[i]=bsm$p[i-1]+(1/(n=1-i))} bsm$p=100*bsm$p/n bsm barplot(ev, main="valeurs propres", col="grey", las=2) abline(h=mean(ev), col="red") legend("topright", "moyenne des valeurs propres", lwd=1, col=2, bty="n")
Cet histogramme montre que la proportion de la variance expliquée par chaque composante chute en-dessous de la proportion moyenne expliquée par l'ensemble des composantes après le sixième axe d“ordination PC6. En consultant le résumé de nouveau, on peut constater que la proportion cumulée de la variance expliquée par les cinq premières composantes principales est de 85%.
La PCA n'est pas seulement appropriée pour les données de composition d'espèces, mais peut également être exécutée sur des variables environnementales standardisées:
- | PCA pour les variables environnementales
#Exécuter la PCA env.pca<-rda(env.z) # ou rda(env, scale=TRUE) #Extraction des résultats summary(env.pca) summary(env.pca, scaling=2)
«Scaling» réfère à quelle portion de la PCA est redimensionnée aux valeurs propres. Scaling = 2 signifie que les scores des espèces sont mises à l'échelle des valeurs propres, alors que scaling = 1 signifie que les scores des sites sont mises à l'échelle des valeurs propres. Scaling = 3 signifie qu'à la fois les scores des espèces et des sites sont mis symétriquement à l'échelle de la racine carrée des valeurs propres. En scaling = 1,les distances euclidiennes entre les sites (lignes de la matrice de données) sont conservées tandis qu'en scaling = 2 les corrélations entre espèces (les colonnes de la matrice de données) sont conservées. Cela implique que lorsque vous regardez un biplot de PCA en Scaling = 2, l'angle entre les descripteurs représente leur corrélation.
- | Axes significatifs
ev<-env.pca$CA$eig ev[ev>mean(ev)] n<-length(ev) bsm<-data.frame(j=seq(1:n), p=0) bsm$p[1]=1/n for (i in 2:n) { bsm$p[i]=bsm$p[i-1]+(1/(n=1-i))} bsm$p=100*bsm$p/n bsm barplot(ev, main="valeurs propres", col="grey", las=2) abline(h=mean(ev), col="red") legend("topright", "moyenne des valeurs propres", lwd=1, col=2, bty="n")
Comparer cet histogramme de valeurs propres avec celui que vous avez créé pour la PCA sur les abondances d'espèces.
Bien que beaucoup d'informations puissent être extraites d'une PCA par la fonction summary de la PCA, l'interpétation et la communication des résultats est souvent facilitée en traçant un biplot. Sur un biplot de PCA, l'axe des x correspond à la première composante principale et l'axe des y à la deuxième composante principale.
La fonction plot () permet de tracer des biplot sur lequels les sites figurent en chiffres noirs et les espèces sont représentées en rouge. Le biplot de la PCA des abondances d'espèces peut être appelé comme suit:
- | Graphique PCA des abondances d'espèces
plot(spe.h.pca)
La construction de biplots de PCA s'articule en trois étapes:
- | Biplot de PCA
plot(spe.h.pca, type=”n”) # Produit une figure vierge points(spe.h.pca, dis=”sp”, col=”blue”) # ajoute les points correspondant aux espèces #utilizer text() plutôt que points() si vous préférez que les codes des espèces s'affichent (nom des colonnes) points(spe.h.pca, dis=”sites”, col=”red”) # ajoute les points correspondant aux sites
Pour créer de plus beaux biplots, essayez ce code:
- | Graphique PCA des abondances d'espèces v.2
#Scaling 1 windows() plot(spe.h.pca) windows() biplot(spe.h.pca) windows() # scaling 1 = distance biplot : # distances entre les objets est une approximation de leur distance euclidienne # les angles entre les descripteurs ne réflètent PAS leur corrélation plot(spe.h.pca, scaling=1, type="none", xlab<-c("PC1 (%)", round((spe.h.pca$CA$eig[1]/sum(spe.h.pca$CA$eig))*100,2)), ylab<-c("PC2 (%)", round((spe.h.pca$CA$eig[2]/sum(spe.h.pca$CA$eig))*100,2))) points(scores(spe.h.pca, display="sites", choices=c(1,2), scaling=1), pch=21, col="black", bg="steelblue", cex=1.2) text(scores(spe.h.pca, display="species", choices=c(1), scaling=1), scores(spe.h.pca, display="species", choices=c(2), scaling=1), labels=rownames(scores(spe.h.pca, display="species", scaling=1)), col="red", cex=0.8)
Le code ci-dessus a produit trois biplots mais le dernier est le plus attrayant :

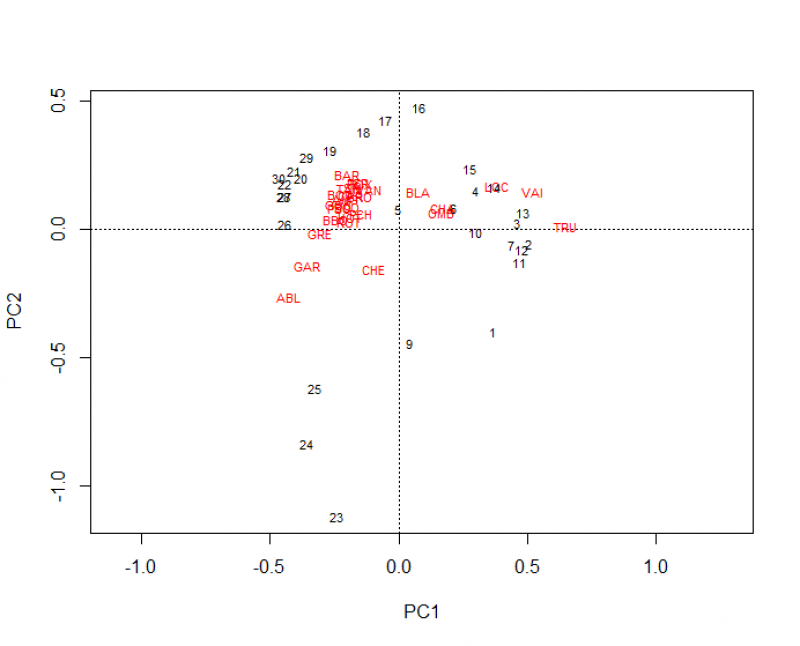
Sur ce graphique, les sites scores sont indiqués par des points bleus et les noms d'espèces sont en rouge. Il est également possible de représenter les sites par leurs noms.
Comment interpréter ce type de graphique ? Ce biplot permet d'observer qu'il existe certains groupes de sites homogènes du point de vue de la composition de leur communautés de poissons. On y voit également que l'espèce «ABL» n'a pas la même prévalence dans la majorité des sites que les autres espèces plus proches du centre du graphique.
Les biplots ne doivent pas seulement être interprétés en termes de proximité, mais également d'angles. Deux variables séparées d'un angle de 90 degrés ne sont pas corrélées. Deux variables très rapprochés sont fortement corrélées. Deux variables aux directions opposées sont corrélées négativement.
Maintenant regardons le biplot de la PCA environnement:
- | Graphique PCA des variables environnementales
#Scaling 2 windows() plot(env.pca) windows() # scaling 2 = graphique de corrélations : # les distances entre les objets ne sont PAS des approximations de leur distance euclidienne # les angles entres les descripteurs reflètent leur corrélation plot(env.pca, scaling=2, type="none", xlab<-c("PC1 (%)", round((env.pca$CA$eig[1]/sum(env.pca$CA$eig))*100,2)), ylab<-c("PC2 (%)", round((env.pca$CA$eig[2]/sum(env.pca$CA$eig))*100,2)), xlim<-c(-1,1), ylim=c(-1,1)) points(scores(env.pca, display="sites", choices=c(1,2), scaling=2), pch=21, col="black", bg="darkgreen", cex=1.2) text(scores(env.pca, display="species", choices=c(1), scaling=2), scores(env.pca, display="species", choices=c(2), scaling=2), labels<-rownames(scores(env.pca, display="species", scaling=2)), col="red", cex=0.8)
Rappelez-vous qu'un biplot de PCA est en fait un nuage de points dans lequel les axes sont des combinaisons linéaires des variables d'origine. Il existe donc beaucoup de façons différentes de tracer un biplot. Par exemple, vous pouvez utiliser la fonction ggplot () et les compétences acquises de l'atelier 3 pour tracer votre graphique d'ordination dans ggplot.
Utilisation des axes de PCA comme variable explicative composite
Dans certains cas, l'utilisateur cherche à réduire un grand nombre de variables environnementales en un plus faible nombre de variables composites. Lorsque les axes de PCA représentent des gradients écologiques (i.e. lorsue les variables environnementales sont corrélées de façon cohérente avec les axes de PCA), l'utlisateur peut utiliser les scores des sites le long axes de PCA dans de nouvelles analyses (au lieu d'utiliser les variables environnementales brutes). En d'autres termes, étant donné que les scores des sites le long des axes de PCA représentent des combinaisons linéaires des descripteurs, ils peuvent être utilisés comme proxy des conditions écologiques dans de nouvelles analyses.
Dans l'exemple ci-dessus, le premier axe de PCA peut être identifié comme un gradient écologique allant des sites oligotrophes riches en oxygène aux sites eutrophes pauvres en oxygène: de gauche à droite, le premier groupe de sites montrent les plus hautes altitudes (alt) et pente (slope), et les plus faibles débit (deb) et distance à la source (das). Le second groupe de sites possède les plus hautes valeurs de concentration en oxygène (oxy) et les plus faibles concentrations en nitrates (nit). Un troisième groupe de sites montrent des valeurs intermédiaire pour l'ensemble de ces variables.
Dans ce cas, si l'objectif est d'identifier si une espèce particulière est associée au gradient oligotrophe-eutrophe, il est possible de corréler l'abondance de cette espèce aux scores des sites le long du premier axe de PCA. Par exemple, si l'utilisateur veut identifier si l'espèce TRU est associée à des eaux oligotrophes ou eutrophes, il lui est possible d’utiliser le modèle linéaire suivant:
- | Graphique PCA des variables environnementales
Sites_scores_Env_Axis1<- scores(env.pca, display="sites", choices=c(1), scaling=2) spe$ANG plot( Sites_scores_Env_Axis1, spe$TRU) summary(lm(spe$TRU~Sites_scores_Env_Axis1)) abline(lm(spe$TRU~Sites_scores_Env_Axis1))
Ce modèle simple montre que l'abondance de l'espèce TRU est significativement liée aux scores des sites le long du premier axe de PCA (t = -5.30, p = 1.35e-05, adj-R2 = 49.22%), c’est-à-dire qu'elle dépend d'un gradient oligotrophe-eutrophe. dans ce cas l'espèce TRU préfère donc les eaux oligotrophes.
Défi 3 Exécuter une PCA sur l'abondance des espèces de mites. Quels sont les axes significatifs ? Quels sont groupes de sites pouvez-vous identifier? Quelles espèces sont liées à chaque groupe de sites?
- | Données mites
mite.spe<-data(mite) # données disponibles dans vegan
Défi - Solution
3.2. Analyse des Correpondances (Correspondence Analysis, CA)
L'une des hypothèses clefs de la PCA postule que les espèces sont liées les unes aux autres de façon linéaire, et qu'elles répondent de façon linéaire aux gradients écologiques. Ce n'est cependant pas nécessairement le cas dans les données écologiques (e.g. beaucoup d'espèces montrent en effet un distribution unimodale le long des gradients environnementaux). Utiliser une PCA avec des données contenant des espèces à distribution unimodale, ou un grand nombre de zéros (absence des espèces), peut conduire à un phénomène statistique appelé “horseshoe effect” (ou effet fer à cheval) se produisant le long de gradients écologiques. Dans de tels cas, l'Analyse des Correspondances (CA) permet de mieux représenter les données (voir Legendre et Legendre pour plus d'informations). Comme la CA préserve les distances de Chi2 entre objets (tandis que la PCA préserve les distances euclidiennes), cette technique est, en effet, plus appropriée pour ordonner les jeux de données contenant des espèces à distribution unimodale, et a, pendant longtemps, était l'une des techniques les plus employées pour analyser les données d'absence-présence ou d'abondances d'espèces. Lors d'une CA, les données brutes sont d'abord transformées en une matrice Q des contributions cellule-par-cellule à la statistique Chi2 de Pearson, puis la matrice résultante est soumise à une décomposition en valeurs singulières afin de calculer les valeurs propres et vecteurs propres de l'ordination.
Le résultat d'une CA représente donc une ordination dans laquelle les distances de Chi2 entre objets sont préservées (au lieu de la distance euclidienne dans une PCA), le distance de Chi2 n'étant pas influencée par la présence de double-zéros. Ainsi, la CA constitue une méthode d’ordination puissante pour l'analyse des abondances brutes d'espèces (i.e. sans pré-transformation). Contrairement à la PCA, la CA peut être appliquée sur des données quantitatives ou binaires (telles que des abondance ou absence-présence d'espèces). Comme dans une PCA, le critère de Kaiser-Guttman peut être utilisé pour identifier les axes significatifs d'une CA, et les scores des objets le long des axes d'ordination peuvent être extraits pour être utlisés dans des régressions multiples par exemple.
Exécuter une CA sur les données d'abondance d'espèces:
- | CA par cca() de vegan
#Effectuer une CA à l'aide de la fonction cca() (NB: cca() est utilisée à la fois pour les CA et CCA) spe.ca <- cca(spe) # Identifier les axes significatifs ev<-spe.ca$CA$eig ev[ev>mean(ev)] n=length(ev) barplot(ev, main="Eigenvalues", col="grey", las=2) abline(h=mean(ev), col="red") legend("topright", "Average eigenvalue", lwd=1, col=2, bty="n")
D’après cet histogramme, à partir du sixième axe d’ordination CA6, la proportion de variance expliquée diminue sous la proportion moyenne expliquée par l'ensemble des axes. La sortie R de la CA ci-dessous montre également que les cinq premiers axes d'ordination explique une proportion cumulée de variance expliquée de 84.63%.
- | Extraire les résultats d'une CA
summary(spe.h.pca) summary(spe.h.pca, diplay=NULL)
Les résultats d'une CA sont présentés sous R de la même façon que ceux d'une PCA. On y observe que le premier axe CA1 explique 51.50% de la variation de l'abondance des espèces tandis que le second axe CA2 explique 12.37% de la variation.
- | Construction des biplots
par(mfrow=c(1,2)) #### scaling 1 plot(spe.ca, scaling=1, type="none", main='CA - biplot scaling 1', xlab=c("CA1 (%)", round((spe.ca$CA$eig[1]/sum(spe.ca$CA$eig))*100,2)), ylab=c("CA2 (%)", round((spe.ca$CA$eig[2]/sum(spe.ca$CA$eig))*100,2))) points(scores(spe.ca, display="sites", choices=c(1,2), scaling=1), pch=21, col="black", bg="steelblue", cex=1.2) text(scores(spe.ca, display="species", choices=c(1), scaling=1), scores(spe.ca, display="species", choices=c(2), scaling=1), labels=rownames(scores(spe.ca, display="species", scaling=1)),col="red", cex=0.8) #### scaling 2 plot(spe.ca, scaling=1, type="none", main='CA - biplot scaling 2', xlab=c("CA1 (%)", round((spe.ca$CA$eig[1]/sum(spe.ca$CA$eig))*100,2)), ylab=c("CA2 (%)", round((spe.ca$CA$eig[2]/sum(spe.ca$CA$eig))*100,2)), ylim=c(-2,3)) points(scores(spe.ca, display="sites", choices=c(1,2), scaling=2), pch=21, col="black", bg="steelblue", cex=1.2) text(scores(spe.ca, display="species", choices=c(1), scaling=2), scores(spe.ca, display="species", choices=c(2), scaling=2), labels=rownames(scores(spe.ca, display="species", scaling=2)),col="red", cex=0.8)
Ces biplots montrent qu'un groupe de sites (à gauche) possède des communautés similaires de poissons caractérisées par de nombreuses espèces dont GAR, TAN, PER, ROT, PSO et CAR; dans le coin supérieur droit, un second groupe de sites se caractérisent par les espèces LOC, VAI et TRU; le dernier groupe de sites dans le coin inférieur droit montrent des communautés abondantes en BLA, CHA et OMB.
Défi 4 Exécuter une CA sur les données d'abondance des espèces d'acariens (données mite). Quels sont les axes importants? Quels groupes de sites pouvez-vous identifier? Quelles espèces sont liées à chaque groupe de sites?
Défi 4 - Solution
3.3 Analyse en coordonnées principales (Principal Coordinates Analysis, PCoA)
La PCA, comme la CA, impose une préservation des distances entre objets: la distance euclidienne dans le cas de la PCA, et la distance de Chi2 dans la CA. Si l'objectif est d'ordonner les objets sur la base d'une autre mesure de distance plus appropriée au problème, la PCoA constitue une technique de choix. Dans une PCA, les données sont pivotées de façon à ce que la première composante principale (correspondant à une combinaison linéaire des descripteurs) explique la plus forte proportion de variation possible; la contribution de chaque descripteur (espèces ou variables environnementales) à chaque composante principale peut alors être évaluée d'après son score. La PCoA est une seconde méthode d'ordination sans contrainte dans laquelle les points sont ajoutés les uns après les autres à l'espace d'ordination en utilisant la distance euclidienne ou n'importe quelle mesure de distance (dissimilarité) métrique vous choisissez. Un premier point est ainsi placé dans l'espèce d'ordination, puis un second point placé à la valeur de distance du premier, puis un troisième et ainsi de suite en ajoutant autant d'axes (de dimensions) que nécessaire. Il est parfois difficile de choisir entre effectuer une PCA ou une PCoA. La PCA permet toutefois de réduire des données multivariables en un faible nombre de dimensions tandis que la PCoA est utile pour visualiser les distances entre sites (ou objets). La PCoA est aussi particulièrement adaptées pour les jeux de données présentant plus de colonnes que de lignes. Par exemple, si des centaines d'espèces ont été observées dans un petit nombree de quadrats, une approche basée sur une PCoA utilisant la distance de Bray-Curtis (voir ci-dessous) peut être plus adaptée.
PCoA avec DoubsSpe (transformé Hellinger):
- | PCoA
# En utilisant la fonction cmdscale() ?cmdscale cmdscale(dist(spe.hel), k=(nrow(spe)-1), eig=TRUE) # En utilisant la fonction pcoa() ?pcoa spe.h.pcoa<-pcoa(dist(spe.hel)) # Extraction des résultats spe.h.pcoa # Représentation graphique biplot.pcoa(spe .h.pcoa, spe.hel, dir.axis2=-1)
Les résultats de cette PCoA sont:
Et le graphique:
Vous pouvez aussi exécuter cette PCoA avec une autre mesure de distance (ex. Bray-Curtis):
- | PCoA avec Bray-Curtis
spe.bray.pcoa<-pcoa(spe.db) # il s'agit de la matrice de distances de Bray-Curtis qu'on a générée plus tôt spe.bray.pcoa biplot.pcoa(spe.bray.pcoa, spe.hel, dir.axis2=-1) # Le choix d'une mesure de distance est très important car ça influence les résultats!
Défi 5 Exécuter une PCoA sur les données d'abondance des espèces d'acariens transformées Hellinger (données mite). Quels sont les axes importants? Quels groupes de sites pouvez-vous identifier? Quelles espèces sont liées à chaque groupe de sites? Comment les résultats de cette PCoA se comparent-ils avec ceux de la PCA?
Défi 5 - Solution
3.4. Positionnement multidimensionnel non-métrique (Nonmetric Multidimensional Scaling, NMDS)
Les méthodes d'ordination non contrainte présentées ci-dessus permettent d'organiser les objets (ex. les sites) caractérisés par des descripteurs (ex. les espèces) dans un espace comprenant l'ensemble des dimensions décrites par l’ellipsoïde représentant le nuage des points de données. En d'autres termes, la PCA, la CA et la PCoA calculent un grand nombre d'axes d'ordination (nombre proportionnel au nombre de descripteurs) représentant la variation des descripteurs entre sites et préservant les distances entre objets (distance euclidienne dans une PCA, distance de Chi2 dans une CA et distance définie par l'utilisateur dans une PCoA). L'utilisateur peut ensuite sélectionner les axes d'intérêt (généralement les deux premiers axes d'ordination) pour représenter les objets dans un biplot. Le biplot produit représente ainsi correctement les distances entre objets (ex. la similarité des sites), mais ne permet pas de représenter l'ensemble des dimensions de la variation dans l'espace d'ordinations (étant donnée que l'Axe 3, l'Axe 4,…, l'Axe n'apparaissent pas sur le biplot, mais contribuent tout de même à expliquer la variation entre objets).
Dans certains cas, la priorité n'est pas de préserver la distance exacte entre les objets, mais au contraire de représenter aussi fidèlement que possible les relations entre objets selon un petit nombre d'axes (généralement deux ou trois) spécifiés par l'utilisateur. Dans de tels cas, le positionnement multidimensionnel non-métrique (NMDS) est la solution. Si l'utilisateur définit un nombre d'axe égal à deux, le biplot produit par le NMDS correspond à la meilleure solution graphique pour représenter en deux dimensions la similarité entre objets (les objets dissimilaires étant les plus éloignées, et les objets similaires étant les plus proches). De plus, le NMDS permet à l'utilisateur de choisir la mesure de distance qu'il souhaite pour ordonner les objets.
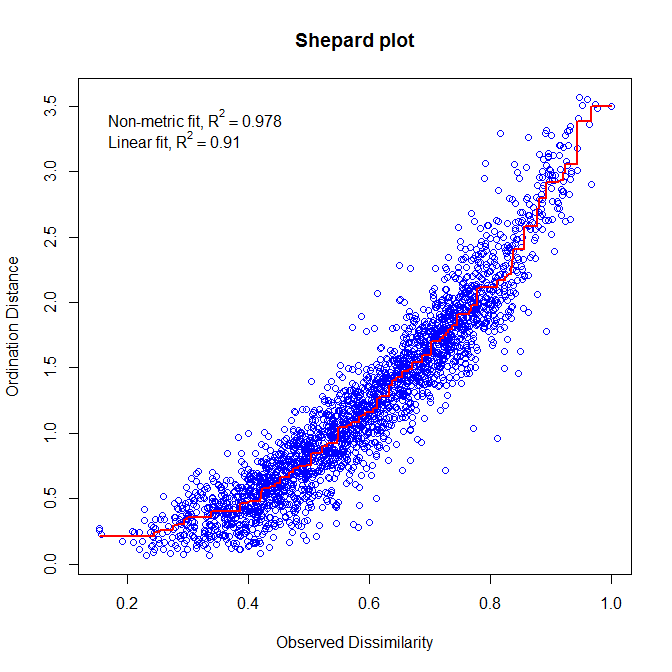
Afin de trouver la meilleure représentation des objets, le NMDS applique une procédure itérative qui vise à positionner les objets dans le nombre spécifié de dimensions de façon à minimiser une fonction de stress (variant de 0 à 1) qui mesure la qualité de l'ajustement de la distance entre objets dans l'espace d'ordination. Ainsi, plus la valeur du stress sera faible, plus la représentation des objets dans l'espace d'ordination sera exacte. Un second moyen d'évaluer l'exactitude d'un NMDS consiste à construire un diagramme de Shepard qui représente les distances entre objets sur le biplot d'ordination en fonction de leurs distances réelles. Le R2 obtenu à partir de la régression entre ces deux types de distance mesure la qualité de l'ajustement du NMDS.
- | NMDS sur les données d’abondance spe avec une distance de Bray-Curtis et k=2 axes d'ordination
# NMDS spe.nmds<-metaMDS(spe, distance='bray', k=2) ### Extraction des résultats spe.nmds ### Évaluation de la qualité de l'ajustement et construction du diagramme de Shepard spe.nmds$stress stressplot(spe.nmds, main='Shepard plot') # Construction du biplot windows() plot(spe.nmds, type="none", main=paste('NMDS/Bray - Stress=', round(spe.nmds$stress, 3)), xlab=c("NMDS1"), ylab=c("NMDS2")) points(scores(spe.nmds, display="sites", choices=c(1,2)), pch=21, col="black", bg="steelblue", cex=1.2) text(scores(spe.nmds, display="species", choices=c(1)), scores(spe.nmds, display="species", choices=c(2)), labels=rownames(scores(spe.nmds, display="species")), col="red", cex=0.8)
Le diagramme de Shepard identifie une forte corrélation entre les distances observées et les distances de l'ordination (R2 > 0.95), et donc une bonne qualité de l'ajustement du NMDS.
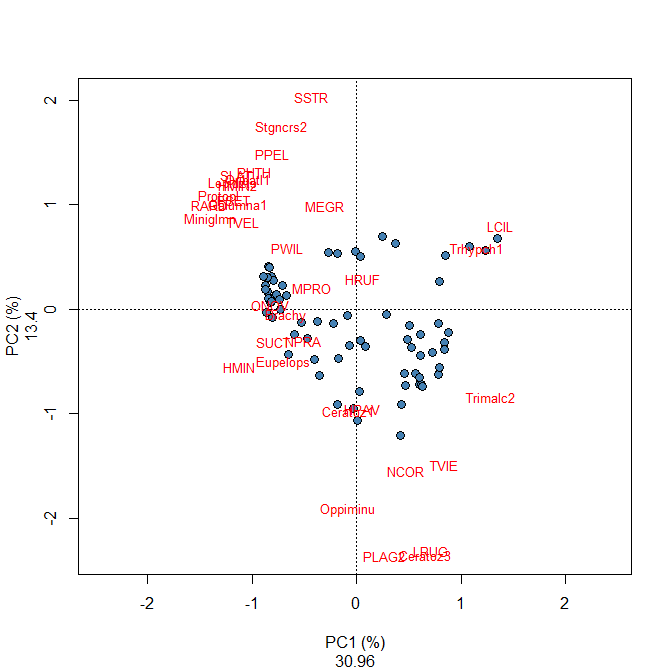
Le biplot du NMDS identifie un groupe de sites caractérisés par les espèces BLA, TRU, VAI, LOC, CHA et OMB, tandis que les autres espèces caractérisent un groupe de sites situés dans le coin supérieur droit du biplot. Quatre sites situés dans le coin inférieur droit sont fortement différents des autres.
Défi 6 Exécuter un NMDS sur les données d'abondance des espèces d'acariens (données mite) en deux dimensions à partir de distances de Bray-Curtis. Évaluer la qualité de l'ajustement et interpréter le biplot.
Défi 5 - Solution
3.5. En résumé
L’ordination constitue une puissante méthode d'analyse pour étudier les relations entre objets caractérisés par différents descripteurs (ex. des sites décrits par leurs communautés biologiques, ou leurs variables environnementales), mais de nombreuse méthodes d'ordination existent. Ces méthodes diffèrent principalement par le type de distance qu'elles préservent, le type de variables qu'elles autorisent, et le nombre de dimensions de l'espace d'ordination. Pour mieux guider votre choix de la méthode d'ordination à utiliser, le tableau ci-dessous identifie les caractéristiques de chacune des quatre méthodes d'ordination présentées lors de cet atelier.
Lors du prochaine atelier, vous verrez comment identifier les relations entre variables environnementales et communautés biologiques décrivant un même ensemble de sites, à l'aide des méthodes d'analyses canoniques.