This is an old revision of the document!
Ateliers R du CSBQ
Cette série de 10 ateliers guide les participants à travers les étapes requises afin de maîtriser le logiciel R pour une grande variété d’analyses statistiques pertinentes en recherche en biologie et en écologie. Ces ateliers en libre accès ont été créés par des membres du CSBQ à la fois pour les membres du CSBQ et pour la grande communauté d’utilisateurs de R.
Le contenu de cet atelier a été révisé par plusieurs membres du CSBQ. Si vous souhaitez y apporter des modifications, veuillez SVP contacter les coordonnateurs actuels de la série, listés sur la page d'accueil
Atelier 3: Introduction à ggplot2, tidyr & dplyr
Développé par : Xavier Giroux-Bougard, Maxwell Farrell, Amanda Winegardner, Étienne Low-Décarie, Monica Granados
Résumé : Pendant cet atelier, vous perfectionnerez vos compétences en visualisation et manipulation de jeux de données à l’aide de trois bibliothèques R : ggplot2, reshape et plyr. D’abord, vous explorerez la richesse de ggplot2 comme solution de rechange aux outils graphiques de base dans R, soit en explorant vos données visuellement et en produisant des figures dignes de publications scientifiques. Ensuite, nous présenterons tidyr et dplyr pour vous aider à transformer et à gérer le format de vos données ainsi que pour appliquer des fonctions simples ou complexes sur des sous-groupes de vos jeux de données. Cet atelier s’avérera utile non seulement pour les participants suivant notre série d’ateliers, mais également pour ceux et celles d’entre vous connaissant déjà le langage R et cherchant à maîtriser des outils plus avancés pour l'analyse et la visualisation de vos données.
Lien vers la présentation Prezi associée : Prezi
Téléchargez le script R pour cet atelier : Script
1. Tracer des graphiques avec R en utilisant la grammaire des graphiques (ggplot2)
Que vous effectuiez des statistiques descriptives (e.g. Excel), des analyses plus avancées (e.g. SAS, JMP, SPSS) ou des graphiques et des tableaux (e.g. Sigmaplot, Excel), il est facile de se perdre dans le flux de travail lorsqu'on utilise plusieurs logiciels. Ceci s'avère particulièrement problématique lorsqu'on doit importer ou exporter des données pour effectuer une tâche en aval. À chaque opération, on augmente le risque d'introduire des erreurs dans les données ou de perdre de vue le “bon” fichier de données. Le langage statistique R fournit une solution à ce problème en regroupant tous les outils nécessaires pour manipuler des données, effectuer des analyses statistiques et produire des graphiques sous un seul logiciel. En regroupant notre flux de travail sous le même toit, on réduit la probabilité de faire des erreurs et on rend notre flux de travail beaucoup plus compréhensible et reproductible. Tout ceci en vaut grandement l'effort!
1.1 Intro - ggplot2
Le paquet ggplot2 offre le cadre graphique le plus flexible et de plus complet pour la visualisation des données dans R. Ce paquet a été développé par Hadley Wickham, qui s'est lui-même basé sur la grammaire des graphiques de Leland Wilkinson. Le code source est hébergé sur github : https://github.com/hadley/ggplot2. Aujourd'hui, nous allons regarder les bases de ggplot2 afin que vous puissiez voir tout le potentiel que ce paquet offre pour la visualisation de données.
Avant de débuter, il faut ouvrir RStudio et charger le paquet ggplot2 :
- |
if(!require(ggplot2)){install.packages("ggplot2")} library(ggplot2)
1.2 Graphiques simples avec la fonction ''qplot()''
Utiliser ''qplot()''
Créons un premier graphique à l'aide de la fonction qplot() du paquet ggplot2. La fonction qplot() (qui signifie “quick plot”) sert de lien intuitif entre la fonction de base plot() et la fonction ggplot() du paquet ggplot2. La syntaxe entre plot() et qplot() est presque identique : ces fonctions peuvent tracer un graphique en fonction du type de données utilisées (e.g. facteurs, variables numériques, etc.). Rappelez-vous que vous pouvez toujours accéder à la page d'aide d'une fonction de la manière suivante :
- |
?qplot
Si vous avez regardé la page d'aide de la fonction qplot(), vous avez probablement remarqué que les trois premiers arguments sont :
- data
- x
- y
- …
Charger les données
Avant d'aller plus loin, il faut charger un jeu de données qui nous permettra de réaliser les exercices de l'atelier. Nous utiliserons le jeu de données iris, un jeu de données bien connu sur les dimensions florales de trois espèces d'iris. Ces données ont été recueillies en Gaspésie par le botaniste américain Edgar Anderson. Le jeu de données est disponible directement dans R. Pour le charger et explorer sa structure, utilisez les commandes suivantes :
Nuage de points
Traçons un premier diagramme de type “nuage de points” de la largeur des sépales en fonction de la longueur des sépales d'iris avec la fonction qplot() :
Nuage de points (variables catégoriques)
Il est également possible de tracer un graphique de type “nuage de points” en utilisant une variable catégorique :
Ajouter des étiquettes d'axes et un titre
En plus des données et des variables à spécifier, on peut inclure plusieurs arguments supplémentaires pour améliorer l'aspect d'un graphique (Référez-vous à la page d'aide.). Commençons par ajouter des étiquettes aux axes ainsi qu'un titre à l'aide des arguments xlab/ylab et main respectivement :
- |
qplot(data = iris, x = Sepal.Length, xlab = "Longueur (mm)", y = Sepal.Width, ylab = "Largeur (mm)", main = "Dimensions de sépales d'iris")
ggplot2 - Défi #1
À l'aide de la fonction qplot(), tracez un graphique de type “nuage de points” avec un titre et des étiquettes d'axes. Utilisez le jeu données CO2 ou BOD qui sont déjà inclus dans R. Vous pouvez les charger et explorer leur structure avec ces commandes :
Solution avec le jeu de données CO2
1.3 La grammaire des graphiques
La grammaire des graphiques est un cadre conceptuel pour la visualisation des données qui permet de décomposer un graphique en plusieurs éléments individuels. L'intérêt majeur de ce cadre est qu'il est possible de modifier chaque élément du graphique séparément. Plusieurs éléments, généralement appelés couches, composent un graphique :
- Éléments esthétiques (aes)
- Objets géométriques (geoms)
- Transformations
- Axes (Système de coordonnées)
- Échelles
Esthétique des graphiques : ''aes()''
Dans ggplot2, les éléments esthétiques sont des paramètres qui spécifient quelles données sont visualisées et comment elle le sont. Voici quelques arguments qui sont utilisés avec la fonction aes() :
x: position des données le long de l'axe des xy: position des données le long de l'axe des ycolour: couleur d'un élémentgroup: groupe auquel un élément appartientshape: forme utilisée pour afficher un pointlinetype: type de ligne utilisée (e.g. continue, en tireté, etc.)size: taille d'un point ou d'une lignealpha: transparence d'un élément
Objets géométriques : ''geoms''
Les objets géométriques ou geoms déterminent la représentation visuelle des données. Voici plusieurs fonctions qui sont utilisées à cette fin :
geom_point(): nuage de pointsgeom_line(): ligne reliant les points par ordre croissant de xgeom_path(): ligne reliant les points par ordre d'apparitiongeom_boxplot(): diagramme de boîte à moustaches pour les variables catégoriquesgeom_bar(): diagramme en bâtons pour les variables catégoriques en axe des xgeom_histogram(): histogramme (commegeom_bar, mais pour une variable continue en axe des x)
Comment ça fonctionne
- Créez un objet simple :
objet.graphique <- ggplot() OU qplot() - Ajoutez des couches :
objet.graphique <- objet.graphique + couche() - Répétez l'étape 2 jusqu'à ce que vous soyez satisfait et faites apparaître le graphique :
print(objet.graphique)
Le graphique final est produit en superposant plusieurs couches jusqu'à l'obtention d'un résultat satisfaisant :
Ressources supplémentaires
Cette brève introduction à la grammaire des graphiques n'est pas suffisante pour couvrir toutes les couches et caractéristiques possibles pour la visualisation des données. Dans cet atelier, nous vous présentons plus bas les couches et caractéristiques les plus utilisées. Voici quelques ressources supplémentaires afin d'explorer l'immense potentiel de ggplot2 et de la grammaire des graphiques :
- Documentation sur ''ggplot2'': ceci est la meilleure ressource sur ggplot2. Elle inclut une liste complète de tous les éléments disponibles ainsi que la liste des arguments nécessaires pour chaque élément en plus d'exemples très utiles.
- SAPE: le groupe de recherche “Software And Programmer Efficiency” consacre une partie de son site internet à l'usage approprié des différents éléments de ggplot2 en plus de présenter des exemples pertinents.
- The Grammar of Graphics: ce livre écrit par Leland Wilkinson présente le cadre conceptuel de la visualisation des données sur lequel se base ggplot2.
- ggplot2: ce livre par Hadley Wickham, l'auteur de ggplot2, montre comment implémenter la grammaire des graphiques dans R.
1.4 Graphiques avancés avec la fonction ''ggplot()''
''qplot()'' vs ''ggplot()''
La fonction qplot() est idéale pour tracer des graphiques rapidement. Tel que mentionné plus haut, si on assigne un graphique qplot à un objet, il est ensuite possible d'ajouter des couches pour augmenter la complexité du graphique : objet.graphique <- objet.graphique + couche(). Cependant, la fonction qplot() ne fait que reprendre les commandes de la fonction ggplot() dans un format similaire à celui de la fonction de base plot(). Regardons la syntaxe sous-jacente de ggplot2 lorsqu'on utilise la fonction qplot().
qplot():
- |
qplot(data = iris, x = Sepal.Length, xlab = "Longueur (mm)", y = Sepal.Width, ylab = "Largeur (mm)", main = "Dimensions de sépales d'iris")
ggplot():
- |
ggplot(data = iris, aes(x = Sepal.Length, y = Sepal.Width)) + geom_point() + xlab("Longueur (mm)") + ylab("Largeur (mm)") + ggtitle("Dimensions de sépales d'iris")
Lorsqu'on utilise la fonction ggplot() directement, on spécifie tout d'abord les données et les variables x et y avec aes(). Ensuite, on ajoute chaque couche une à la fois. Cette façon de faire permet d'utiliser le plein potentiel de la grammaire des graphiques. Il est préférable d'utiliser la fonction ggplot() lorsqu'on dois tracer des graphiques plus complexes. C'est ce que nous ferons pour le reste de cet atelier.
Attribuer un graphique à un objet
Avant d'aller plus loin avec les fonctions avancées, créons un objet graphique de base en assignant le graphique précédent à un objet :
1.5 Ajouter des couleurs et des formes
L'argument colour de la fonction aes() permet d'ajouter des couleurs. L'argument shape permet d'attribuer un symbole unique à chaque groupe :
- |
graph.base <- graph.base + aes(colour = Species, shape = Species) graph.base
1.6 Ajouter des objets géométriques
Nous avons déjà des objets géométriques dans notre graphique de base (i.e. des points ajoutés avec la fonction geom_points()). Ajoutons maintenant des lignes de régression avec la fonction geom_smooth() :
- |
graph.ligne <- graph.base + geom_smooth(method="lm", se = FALSE) graph.ligne
BONUS
Il est même possible d'utiliser des émoticônes comme objets géométriques dans vos graphiques!!! Vous aurez tout simplement besoin du paquet emojiGG développé par David Lawrence Miller, disponibles depuis le répertoire GitHub:
- |
devtools::install_github("dill/emoGG") library(emoGG) # vous devez faire une recherche emoji_search("bear") 830 bear 1f43b animal 831 bear 1f43b nature 832 bear 1f43b wild ggplot(iris, aes(Sepal.Length, Sepal.Width, color = Species)) + geom_emoji(emoji="1f337")
ggplot2 - Défi #2
Créez un graphique coloré avec une droite de régression à partir des jeux de données CO2 ou msleep de R :
Solution avec le jeu de données CO2
1.7 Séparer les graphiques en panneaux
La visualisation des données peut être difficile lorsque plusieurs facteurs sont inclus dans une expérience. Par exemple, le jeu de données CO2 contient des données d'absorption de CO2 pour des plantes refroidies et des plantes non refroidies dans deux régions différentes (Québec et Mississippi). Commençons par créer un graphique de base :
- |
data(CO2) CO2.graph <- ggplot(data = CO2, aes(x = conc, y = uptake, colour = Treatment)) + geom_point() + xlab("Concentration en CO2 (mL/L)") + ylab("Absorption de CO2 (umol/m^2 sec)") + ggtitle("Absorption de CO2 par une espèce de graminée") CO2.graph
Si on veut comparer les résultats d'absorption par région, ça peut être utile de représenter les données dans deux panneaux où les axes sont alignés afin de faciliter la comparaison. Avec ggplot2, la fonction facet_grid() permet de découper le graphique en différents groupes. La syntaxe générale est décrite ainsi : plot.object + facet_grid(lignes ~ colonnes). Ici, lignes et colonnes sont des facteurs du tableau de données qu'on souhaite comparer en séparant les données par panneau. Pour comparer les deux régions, on peut utiliser le script suivant :
- |
CO2.graph <- CO2.graph + facet_grid(. ~ Type) CO2.graph
Note : Il est possible de tracer les panneaux verticalement facet.grid(Type ~ .).
1.8 Ajouter des groupes
On peut ajouter des droites de régression de l'abosorption du CO2 en fonction de la concentration en CO2 pour chaque région à l'aide de la fonction geom_line() :
- |
CO2.graph + geom_line()
Les points sont reliés verticalement pour chaque niveau de concentration en CO2. Si on regarde plus en détail la structure du jeu de données CO2, on note que chaque niveau de concentration est répété trois fois par région. Si on veut tracer une droite pour chaque répétition, il faut ajouter l'élément esthétique group à la fonction “geom_line()” :
- |
CO2.graph <- CO2.graph + geom_line(aes(group = Plant)) CO2.graph
ggplot2 - Défi #3
Familiarisez-vous avec un nouvel objet géométrique et d'autres éléments graphiques. Utilisez votre propre jeu de données ou un jeu de données déjà inclus dans R (Tapez data() pour la liste des jeux de données disponibles). Regardez cette page pour un peu d'inspiration !
- |
data(msleep) data(OrchardSprays)
Exemple avec le jeu de données "OrchardSprays"
1.9 Enregistrer un graphique
Plusieurs options s'offrent à vous lorsque vient le temps d'enregistrer un graphique.
Enregistrer un graphique avec RStudio
Avec RStudio, plusieurs options sont disponibles pour enregistrer les graphiques. Vous pouvez les copier dans le presse-papiers ou les exporter vers à peu près n'importe quel type de fichier graphique (png, jpg, emf, tiff, pdf, metafile, etc.) :
Enregistrer un graphique avec un script
Lorsque que vous devez reproduire plusieurs graphiques (e.g. lors d'une analyse qui crée plusieurs graphiques automatiquement), c'est utile d'enregistrer tous ces graphiques dans un seul fichier pdf. La commande suivante permet de réaliser ceci :
Enregistrer un graphique - autres options
There are many other options, of particular note is ggsave() as it will write directly to your working directory all in one line of code and you can specify the name of the file and the dimensions of the plot:
Il existe encore plusieurs autres options, mais parmi celles-ci, ggsave() est particulièrement utile. Cette fonction nous permet d'enregistrer nos graphiques sous multiples formats graphiques, en plus de nous permettre de spécifier les dimensions exactes du produit final:
1.10 Ajustement de précision - couleurs
Le paquet ggplot2 choisit automatiquement les couleurs d'un graphique en se basant sur le cercle chromatique et le nombre de couleurs nécessaires pour réaliser le graphique. Cependant, si vous désirez spécifier vous-même les couleurs à utiliser, vous devez inclure la fonction scale_colour_manual() dans votre script :
Pour un contrôle manuel encore plus précis, vous pouvez spécifier les codes hexadécimaux des couleurs à utiliser.
Un paquet particulièrement simple et utile pour les couleurs s'agit de viridis. Celui-ci offre 4 palettes de couleurs développée pour les visulalizations cartographiques. Les 4 palettes offrent des contrastes riches, et conservent leur informations lorsqu'elles sont converties sur échelle noir-blanc. Voici un example:
- |
CO2.graph + scale_colour_manual(values = viridis(2, option = "D"))
BONUS
Allez jeter un coup d'oeil aux liens/paquets suivants pour plus d'options de couleurs :
- Cookbook for R: cette section du “Cookbook for R” sur ggplot2 présente beaucoup d'idées et de chartes de couleurs.
- RColorBrewer: ce paquet de R est rempli de gradients de couleurs pouvant être intégrés à ggplot2 avec la fonction
scale_color_brewer(). - WesAnderson: ce paquet de R, disponible via GitHub, est rempli de thématiques de couleurs basées sur les films de Wes Anderson !
1.11 Ajustement de précision - axes et échelles
Il est possible de contrôler l'aspect des axes et les échelles utilisées pour représenter les données (e.g. étendue de l'axe, positions, etc.) :
1.12 Ajustement de précision - thèmes
Les thèmes permettent de créer des graphiques et des figures de très haute qualité. Les thèmes permettent de contrôler les éléments esthétiques qui ne sont pas directement reliés aux données (e.g. couleur d'arrière-plan, type de police, format de la légende, etc.).
L'arrière-plan gris par défaut ne vous enchante peut-être pas… Rassurez-vous ! Comme n'importe quel autre aspect dû à la grammaire des graphiques, vous pouvez modifier l'allure d'un graphique selon vos besoins ou votre sens de l'esthétisme. Voici comment faire :
- |
objet.graph + theme()
Il existe une très grande quantité de thèmes inclus dans le paquet ggplot2; vous trouverez la liste complète ici. Vous n'êtes pas obligés de spécifier tous les éléments contenus dans la fonction theme() : vous pouvez débuter avec un thème déjà établi (i.e. avec des paramètres de départ). Par exemple, on peut utiliser le thème “noir et blanc” de la manière suivante :
- |
CO2.graph + theme_bw() # L'arrière-plan est devenu blanc !
Créer son propre thème
Il est très facile de créer son propre thème. Ça peut être très pratique pour produire des figures selon un format précis (e.g. pour une revue scientifique). Le plus simple est de partir d'un thème existant et de modifier ses paramètres :
Le paquet ggtheme
Le paquet ggtheme est un projet intéressant développé par Jeffrey Arnold. Vous savez comment l'installer :
- |
install.packages('ggthemes', dependencies = TRUE) library(ggthemes)
Ce paquet contient plusieurs thèmes, objets géométriques et gradients de couleurs adaptés pour ggplot2. Plusieurs éléments sont basés sur les travaux de personnes influentes dans le domaine de la visualisation des données tel qu'Edward Tufte ou sur les publications des journalistes et programmeurs du blog FiveThirtyEight.
Voici un petit exemple rapide qui utilise les diagrammes de boîte à moustaches de Tufte. Comme vous pouvez le constater, c'est très minimaliste :
- |
data(OrchardSprays) tufte.box.plot <- ggplot(data = OrchardSprays, aes(x = treatment, y = decrease)) + geom_tufteboxplot() + theme_tufte() print(tufte.box.plot)
1.13 ggplot avec un interface utilisateur graphique
Il n'y a aucun mal à utiliser une interface utilisateur graphique pour produire des figures ! Jeroen Schouten, un programmeur de très haut niveau, a rapidement compris que la courbe d'apprentissage de R peut être très à pic pour les débutants. C'est pourquoi il a créé une interface utilisateur graphique en ligne pour ggplot2. Ce n'est peut-être pas aussi efficace que de coder directement les principes de la grammaires des graphiques, mais c'est quand même très complet. Vous pouvez importer des données provenant d'Excel, Google Spreadsheets ou de tout autre format et créer des figures en vous basant sur les tutoriels disponibles. Un aspect intéressant est que vous pouvez voir le code utilisé une fois le graphique réalisé. Vous pouvez donc copier-coller ce code dans R et l'utiliser pour générer des graphiques plus complexes (e.g. incluant différents thèmes).
2. Utiliser "tidyr" pour réorganiser ses données
2.1 Pourquoi réorganiser ses données ?
La réorganisation permet de modifier la structure des données tout en préservant l'information contenue dans le jeu de données. Plusieurs fonctions dans R exigent ou fonctionnent mieux avec une structure de données qui n'est pas nécessairement propice à la lecture par l'oeil humain.
En comparaison à l'agrégation, où plusieurs cellules peuvent être regroupées pour créer un nouveau jeu de données (e.g. tableau des moyennes de plusieurs variables), la réorganisation maintient le même nombre de cellules. Le paquet tidyr permet de réorganiser nos jeu de données dans un format idéal pour nos tâches en aval, et ce à l'aide d'une syntaxe simple et logique.
Commençons par installer et charger le paquet tidyr :
- |
if(!require(tidyr)){install.packages("tidyr")} library(tidyr)
Jeux de données "airquality"
En plus du jeu de données CO2, nous allons utiliser le jeu de données airquality pour cette section de l'atelier.
Explorez la structure du jeu de données airquality:
Vous pouvez faire apparaître la liste des jeux de données disponibles dans R avec la commande suivante : data().
2.2 Données en format long vs large
Supposons que vous envoyez votre assistant de terrain pour faire la collecte de données des dimensions de plusieurs arbres sur un site de recherche, sois le diamètre à la hauteur de la poitrine (DHP) et la hauteur. Il ou elle vous revient avec le format “dégât” (format en large) suivant :
- |
> degat <- data.frame( Species = c("Chêne", "Orme", "Frêne"), DHP = c(12, 20, 13), Haut = c(56, 85, 55)) > degat Species DHP Haut 1 Chêne 12 56 2 Orme 20 85 3 Frêne 13 55
En contraste à l'exemple ci-haut, un jeu de données en format “long” contient une colonne spécifiant les variables mesurées et une autre colonne contenant les mesures correspondantes (chaque colonne est une variable, et chaque rangée s'agit d'une observation unique). En ce sens, les jeux de données en “long” sont plus “propre” (tidy en anglais), parce qu'ils sont plus facilement interprétés par R pour nos visualisations et nos analyses.
Le format de vos données dépend de vos besoins d'analyse et de visualisation, mais des fonctions et des paquets tels que ggplot2 fonctionnent bien avec des données en format long.
De plus, les données en format long peuvent être agrégées et réorganisées en format large plus facilement afin de produire des résumés ou de vérifier si un jeu de données est équilibré (i.e. avec le même nombre d'observations par traitement).
On utilise le paquet tidyr pour :
- 1. “ramasser” les données (gather data en anglais; large –> long)
- 2. “éparpiller” les données (spread data en anglais; long –> large)
Pour vous rappeler de ces deux termes, pensez au ménage chez soi: lorsqu'on éparpille nos données (en format large) c'est le dégât, tandis que lorsqu'on passe le balai, on ramasse les données pour les empiler de manières propres (en format long).
2.3 Gather: Ramasser les données en format long
- |
?gather
La plupart des paquets dans le “Hadleyverse” requièrent un format “long”, c'est-à-dire où chaque rangée est une observation unique, et chaque colonne est une variable. Utilisons la fonction gather() pour “ramasser” nos données. gather() prend plusieurs colonnes et les empile dans deux colonnes: une colonne spécifiant la variable mesurée, et l'autre spécifiant la mesure associée. Il faut inclure les arguments suivant dans la fonction :
- le jeu de données
- le nom de la nouvelle colonne spécifiant la variable mesurée
- le nom de la nouvelle colonne spécifiant la mesure associée
- les colonnes qu'on veut empiler dans le jeu de données
Voici une démonstration avec le jeu de données degat contenant les dimensions d'arbres:
- |
> degat.long <- gather(degat, dimension, cm, c(DHP, Haut)) > degat.long Species dimension cm 1 Chêne DHP 12 2 Orme DHP 20 3 Frêne DHP 13 4 Chêne Haut 56 5 Orme Haut 85 6 Frêne Haut 55
Essayons également avec le jeu de données CO2, soit en “ramassant” les colonnes contant la concentration de CO2 (conc) et le CO2 absorbé (uptake) :
2.4 Spread: Éparpiller les données en format large
- |
?spread
La fonction spread() s'agit de l'homologue inverse de gather(), donc elle nous permet de transformer nos données en format large. On utilise donc la même syntaxe:
- |
> degat.large <- spread(degat.long, dimension, cm) > degat.large Species DHP Haut 1 Chêne 12 56 2 Frêne 13 55 3 Orme 20 85
tidyr - Défi #4
Réorganisez le jeu de données airquality en format long. Ensuite, remettez-le en format large pour retrouver le format original de airquality.
2.5 Separate : séparer une colonne en plusieurs colonnes
Il arrive parfois de travailler avec des jeux de données contenant une ou plusieurs variables dans la même colonne. Dans ces situations, la fonction separate() offre une solution facile pour séparer les variables pour que chacune d'entre elle ait sa propre colonne.
À titre d'exemple, créons un jeu de données fictif sur les poissons et le zooplancton :
- |
set.seed(8) gros.degat <- data.frame(id = 1:4, trt = sample(rep(c('controle', 'culture'), each = 2)), zooplancton.T1 = runif(4), poisson.T1 = runif(4), zooplancton.T2 = runif(4), poisson.T2 = runif(4)) id trt zooplancton.T1 poisson.T1 zooplancton.T2 poisson.T2 1 1 controle 0.3215092 0.76914695 0.4323914 0.001301721 2 2 controle 0.7189275 0.64449114 0.5449621 0.264458864 3 3 culture 0.2908734 0.45704489 0.1382243 0.276532247 4 4 culture 0.9322698 0.08930101 0.9278123 0.521107042
Dans ce cas, notre première étape consisterait à “ramasser” le jeu de données pour le transformer en format long.
- |
> gros.degat.long <- gather(gros.degat, taxa, count, -id, -trt) > head(gros.degat.long) id trt taxa count 1 1 controle zooplancton.T1 0.3215092 2 2 controle zooplancton.T1 0.7189275 3 3 culture zooplancton.T1 0.2908734 4 4 culture zooplancton.T1 0.9322698 5 1 controle poisson.T1 0.7691470 6 2 controle poisson.T1 0.6444911
Petit rappel rapide, ici nous utilisons -id et -trt, ce qui est interprété par la fonction comme étant TOUT sauf les colonnes id et trt. Nous pourrions également énumeré chaque colonne comme ceci : c(zooplancton.T1, poisson.T1, zooplancton.T2, poisson.T2). Quoique le résultat final est identique, l'argument serait très long.
Maintenant, supposons que nous voulons une colonne séparée contenant uniquement la variable du temps (T1 et T2) pour nos analyses. La fonction separate() nous permet de séparer le contenu d'une colonne en utilisant les arguments suivant:
data: le jeu de donnéescol: le nom de la colonne que nous voulons séparerinto: les noms des 2 nouvelles colonnesby: le critère expliquant où/comment séparer le contenu
Dans notre exemple, nous voulons séparer le contenu de la colonne au point (.). Pour le dernier argument dans la fonction separate(). Il faut donc utiliser un petit truc nous provenant des outils d'expressions régulières pour travailler avec les chaînes de caractères (strings en anglais) :
- |
> gros.degat.long.sep <- separate(gros.degat.long, taxa, into = c("especes", "temps"), sep = "\\.") > head(gros.degat.long.sep) id trt especes temps count 1 1 controle zooplancton T1 0.3215092 2 2 controle zooplancton T1 0.7189275 3 3 culture zooplancton T1 0.2908734 4 4 culture zooplancton T1 0.9322698 5 1 controle poisson T1 0.7691470 6 2 controle poisson T1 0.6444911
Ici, notons que la syntaxe \\. est requise parce qu'un point seul (.) est une carte frime (un joker) pour plusieurs fonctions dans le langage R. Les \\ servent donc à indiquer qu'on fait belle et bien référence au caractère ..
2.6 Combiner ggplot2 avec tidyr
Exemple montrant comment utiliser les formats long et large avec le jeu de données "airquality"
- |
head(airquality) Ozone Solar.R Wind Temp Month Day 1 41 190 7.4 67 5 1 2 36 118 8.0 72 5 2 3 12 149 12.6 74 5 3 4 18 313 11.5 62 5 4 5 NA NA 14.3 56 5 5 6 28 NA 14.9 66 5 6
Le jeu de données est en format large où chaque variable mesurée (ozone, solar.r, wind and temp) ont leur propre colonne.
Diagrammes de diagnostic avec un format large et ggplot2
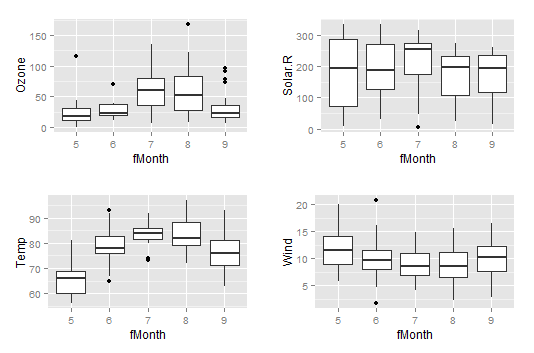
1: Visualisez l'étendue de chaque variable mesurée en fonction du mois.
- |
fMonth <- factor(airquality$Month) # Convertit la variable "Month" en facteur. ozone.box <- ggplot(airquality, aes(x = fMonth, y = Ozone)) + geom_boxplot() solar.box <- ggplot(airquality, aes(x = fMonth, y = Solar.R)) + geom_boxplot() temp.box <- ggplot(airquality, aes(x = fMonth, y = Temp)) + geom_boxplot() wind.box <- ggplot(airquality, aes(x = fMonth, y = Wind)) + geom_boxplot()
Vous pouvez utiliser la fonction grid.arrange() du paquet gridExtra pour regrouper tous ces diagrammes en une seule figure :
- |
combo.box <- grid.arrange(ozone.box, solar.box, temp.box, wind.box, nrow = 2) # nrow = argument spécifiant le nombre de lignes sur lesquelles les diagrammes seront affichés.
On obtient quatre diagrammes distincts sur un seul panneau. Prenez note que l'axe des y est différent pour chaque variable :
2. On peut faire la même chose, mais en représentant les variables en fonction du jour pour chaque mois séparément :
- |
ozone.plot <- ggplot(airquality, aes(x = Day, y = Ozone)) + geom_point() + geom_smooth() + facet_wrap(~Month, nrow = 2) solar.graph <- ggplot(airquality, aes(x = Day, y = Solar.R)) + geom_point() + geom_smooth() + facet_wrap(~Month, nrow = 2) wind.graph <- ggplot(airquality, aes(x = Day, y = Wind)) + geom_point() + geom_smooth() + facet_wrap(~Month, nrow = 2) temp.graph <- ggplot(airquality, aes(x = Day, y = Temp)) + geom_point() + geom_smooth() + facet_wrap(~Month, nrow = 2)
On peut également regrouper tous ces diagrammes (même si ce n'est pas très joli pour le moment !) :

MAIS, comment faire pour utiliser la fonction facet_wrap() avec des variables mesurées au lieu des variables “Month” et “Day” ?
Il faut réorganiser les données en format long ! (Voir la section 2.3)
- |
air.long <- gather(airquality, variable, value, -Month, -Day) air.large <- spread(air.long , variable, value)
Utilisez air.long:
- |
fMonth.long <- factor(air.long$Month) meteo <- ggplot(air.long, aes(x = fMonth.long, y = value)) + geom_boxplot() + facet_wrap(~variable, nrow = 2) meteo
Comparez les diagrammes meteo avec ceux de combo.box :
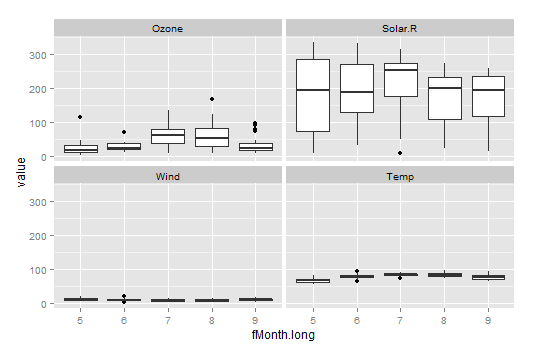
Dans chacun des cas, on a utilisé les mêmes données, mais les graphiques sont légèrement différents.
Les variables de l'objet meteo sont sur la même échelle en utilisant la fonction facet_wrap().
Ça peut être utile dans plusieurs circonstances, mais ça ne permet pas de voir toute la variation de la variable “Wind”.
Dans ce cas, on peut modifier le script pour permettre à R de déterminer l'échelle pour chaque variable avec l'argument scales :

On peut aussi utiliser le format long pour créer un graphique qui inclut toutes les variables sur un seul panneau :
- |
meteo2 <- ggplot(air.long, aes(x = Day, y = value, colour = variable)) + geom_point() + facet_wrap(~Month, nrow = 1) meteo2

3. Manipulation de données avec dplyr
3.1 Intro - la mission de dplyr
Souvent, on doit faire appel à une gamme d'outils complexes pour manipuler nos jeux de données. La mission de dplyr est de simplifier nos tâches de manipulation en distillant toutes les opérations communes sous un même toit. Le résultat est une collection de fonctions ayant une syntaxe simple qu'on peut exécuter à l'aide de verbes intuitifs. Le pont entre nos pensées/intentions et le langage R est donc franchit avec plus de fluidité. En plus d'être facile à utiliser, le paquet dplyr est exceptionnel pour les raisons suivantes :
- il peut traiter des gros jeux de données très rapidement (écrit en langage
Cpp) - sa fonctionnalité est bien intégrée dans RStudio et autres paquets du Hadleyverse
- il peut établir des connections à des bases de données SQL, et traduire votre code en requête SQL
- c'est le Batman des paquets
R(maîtrise la peur des données et adopte des technologies avancées)
3.2 Fonctions dplyr de base
Au coeur du paquet dplyr, on retrouve des “verbes” essentiels qui nous permettent d'accomplir la majeure partie de nos opérations de manipulation de données. Voici 4 verbes qui exécutent les opérations les plus commune :
select(): sélectionne des colonnes dans un jeu de donnéesfilter(): filtre des rangées suivant les critères spécifiésarrange(): trie les données d'une colonne en ordre croissant ou décroissantmutate(): crée des données dans une nouvelle colonne (ou transforme une colonne existante)
Commençons par installer et charger le paquet dplyr afin d'explorer ces fonctions :
- |
if(!require(dplyr)){install.packages("dplyr")} library(dplyr)
Dans les exemples et les défis qui suivent, nous utiliserons les jeux de données airquality et ChickWeight :
Sélection de colonnes avec ''select()''
Le jeu de données airquality contient plusieurs colonnes :
- |
> head(airquality) Ozone Solar.R Wind Temp Month Day 1 41 190 7.4 67 5 1 2 36 118 8.0 72 5 2 3 12 149 12.6 74 5 3 4 18 313 11.5 62 5 4 5 NA NA 14.3 56 5 5 6 28 NA 14.9 66 5 6
À titre d'exemple, supposons qu'on s'intéresse à la variation de la variable Ozone avec le temps. À l'aide de la fonction select(), on peut retenir uniquement les colonnes requises pour nos analyses :
- |
> ozone <- select(airquality, Ozone, Month, Day) > head(ozone) Ozone Month Day 1 41 5 1 2 36 5 2 3 12 5 3 4 18 5 4 5 NA 5 5 6 28 5 6
Comme vous pouvez voir, la syntaxe générale de la fonction est comme suit : select(donnés, colonne1, colonne2, …). La plupart des fonctions dplyr sont exécutées avec une syntaxe simple similaire.
Sélection d'un sous-ensemble de rangées avec ''filter()''
Souvent on s'intéresse à un sous-ensemble spécifique de notre jeu de données. Par exemple, supposons qu'on s'intéresse aux périodes de canicules du mois d'août dans le jeu de données airquality :
- |
> aout <- filter(airquality, Month == 8, Temp >= 90) > head(aout) Ozone Solar.R Wind Temp Month Day 1 89 229 10.3 90 8 8 2 110 207 8.0 90 8 9 3 NA 222 8.6 92 8 10 4 76 203 9.7 97 8 28 5 118 225 2.3 94 8 29 6 84 237 6.3 96 8 30
Ici, le format général de la syntaxe utilisé est comme suit : filter(dataframe, proposition logique 1, proposition logique 2, …). On se rappelle que la réponse à une proposition logique est VRAI ou FAUX (TRUE or FALSE). La fonction filter() retient uniquement les rangées pour lesquelles la réponse à la proposition logique est VRAI. On peut aussi utiliser cette fonction sur des chaînes de caractères (strings) et des facteurs (factors).
Triage avec ''arrange()''
Parfois on travaille avec des jeux de données qui doivent être analysé et/ou visualisé dans un ordre spécifique (e.g. série temporelle). Avec la fonction arrange(), on peut trier le contenu d'une colonne en ordre croissant ou décroissant (alphabétiquement ou numériquement). Commençons par créer une version de airquality dans un ordre aléatoire :
- |
> air_degat <- sample_frac(airquality, 1) > head(air_degat) Ozone Solar.R Wind Temp Month Day 21 1 8 9.7 59 5 21 42 NA 259 10.9 93 6 11 151 14 191 14.3 75 9 28 108 22 71 10.3 77 8 16 8 19 99 13.8 59 5 8 104 44 192 11.5 86 8 12
Maintenant, voici comment réarranger le jeu de donné en ordre chronologique, soit en ordre croissant de Month et ensuite en ordre croissant de Day :
- |
> air_chron <- arrange(air_degat, Month, Day) > head(air_chron) Ozone Solar.R Wind Temp Month Day 1 41 190 7.4 67 5 1 2 36 118 8.0 72 5 2 3 12 149 12.6 74 5 3 4 18 313 11.5 62 5 4 5 NA NA 14.3 56 5 5 6 28 NA 14.9 66 5 6
Notez qu'on peut également réarranger en ordre décroissant en enveloppant la variable d'intérêt avec la fonction desc() au sein de la fonction arrange().
Créer des nouvelles variables avec ''mutate()''
Au delà de l'extraction de sous-ensemble ou du triage, une des opérations les plus communes et les plus utiles s'agit de transformer nos variables existantes afin de générer des nouvelles variables. Par exemple, dans le jeu de données airquality la température est en degré Fahrenheit. Transformons la variable Temp en degré Celsius :
- |
> airquality_C <- mutate(airquality, Temp_C = (Temp-32)*(5/9)) > head(airquality_C) Ozone Solar.R Wind Temp Month Day Temp_C 1 41 190 7.4 67 5 1 19.44444 2 36 118 8.0 72 5 2 22.22222 3 12 149 12.6 74 5 3 23.33333 4 18 313 11.5 62 5 4 16.66667 5 NA NA 14.3 56 5 5 13.33333 6 28 NA 14.9 66 5 6 18.88889
Notez que dans notre exemple, la syntaxe est très simple. Cependant, à l'intérieur d'un seul appel de la fonction mutate() on peut:
- remplacer le contenu de colonnes existantes (en conservant le même nom)
- créer plusieurs nouvelles colonnes
- créer des nouvelles colonnes calculées depuis des nouvelles variables créés en amont
3.3 dplyr et magrittr, don du ciel
Le paquet magrittr introduit un nouvel outil commun dans plusieurs langages informatiques : le pipe. Le “pipe” (un tuyau en français) permet de relier nos fonctions en redirigeant la sortie (output) d'une fonction en amont vers l'entrée (input) d'une fonction en aval. Le “pipe” fournit par le paquet magrittr s'écrit come ceci : %>%. Lorsqu'on l'utilise en combo avec dplyr, le “pipe” de magrittr nous permet d'accéder à la flexibilité et l'omnipotence du paquet. Pour vous familiariser avec ce type de flux de travail, nous utiliserons dplyr avec magrittr pour le reste de l'atelier. Installons et chargeons le paquet :
- |
if(!require(magrittr)){install.packages("magrittr")} library(magrittr)
L'utilisation de magrittr est très intuitif. Nous allons démontrer son fonctionnement en combinant les exemples ci-haut. Supposons qu'on veut créer un sous-ensemble de airquality pour le mois de juin, et ensuite convertir la variable de la température en degré Celsius. Si on procède comme auparavant, soit une étape à la fois, notre code lirait comme suit :
- |
juin_C <- mutate(filter(airquality, Month == 6), Temp_C = (Temp-32)*(5/9))
Ce code peut être difficile à lire parce que l'ordre des opérations exécutées commence au centre et se lit vers l'extérieur jusqu'à la fonction enveloppant le tout. Plus on ajoute des opérations, plus le code devient illisible. Au lieu d'envelopper tous les fonctions, on peut écrire les opérations en ordre d'exécutions et les relier à l'aide du “pipe” %>% :
- |
juin_C <- airquality %>% filter(Month == 6) %>% mutate(Temp_C = (Temp-32)*(5/9))
Notez qu'au sein de chaque fonction, nous avons omis le premier argument spécifiant le jeu de données. Au lieu, nous inscrivons le jeu de données avant les opérations et on le “pipe” jusqu'à la prochaine fonction à l'aide de %>%. Rappelez-vous que cette approche est semblable à ggplot2, puisque nous spécifions le jeu de données à tracer une seule fois, et chaque “couche” supplémentaire est ajouté avec un + pour relier l'information. En utilisant le “pipe”, le code est moins redondant. De plus, il se lit et s'écrit dans le même ordre que l'exécution des opérations, ce qui facilite/accélère la traduction de nos pensées en code ainsi que la lecture et la compréhension du code écrit par nos collègues. Lorsque nos opérations de manipulation de données deviennent plus complexes et requièrent plusieurs étapes, on s'aperçoit rapidement que magrittr offre une approche puissante et élégante pour la rédaction de notre code dplyr.
BONUS : Dans RStudio, on peut insérer le “pipe” rapidement avec la combinaison des touches suivantes sur vos claviers : Ctrl (or Cmd sur Mac) +Shift+M.
3.4 dplyr - Opérations de regroupement et d'aggrégation
Les verbes dplyr couvert jusqu'à prsent sont certes utiles individuellement, mais ils deviennent particulièrement puissant lorsqu'on les relie ensemble avec le “pipe” (%>%) et lorsqu'on les applique sur des sous-groupes d'observations. Les fonctions dplyr suivantes nous permettent donc de séparer nos jeu de données en groupes distincts sur lesquels on peut exécuter des opérations individuelles, comme des fonctions d'aggrégation :
group_by(): regrouper le jeu de donner par un facteur pour les opérations en aval (commesummarise)summarise(): créer un sommaire de variables au sein de groupes distincts dans un jeu de données en utilisant des fonctions d'aggrégation (e.g.min(),max(),mean(), etc…)
Ces deux verbes fournissent la structure requise pour la stratégie Split-Apply-Combine (Séparer-Appliquer-Combiner) originalement introduite dans le paquet plyr, l'ancêtre de dplyr. Voici un exemple de l'usage de ces deux fonctions à l'aide du jeu de données airquality. Supposons qu'on s'intéresse à la température moyenne et l'écart type pour chaque mois:
- |
> mois_moy <- airquality %>% group_by(Month) %>% summarise(mean_temp = mean(Temp), sd_temp = sd(Temp)) mois_moy Source: local data frame [5 x 3] Month mean_temp sd_temp (int) (dbl) (dbl) 1 5 65.54839 6.854870 2 6 79.10000 6.598589 3 7 83.90323 4.315513 4 8 83.96774 6.585256 5 9 76.90000 8.355671
dplyr Défi #5
En utilisant le jeu de données ChickWeight, créez un tableau sommaire dans lequel on retrouve la différence de masse entre le maximum et le minimum de la masse enregistré pour chaque poussin dans l'étude. Utilisez les verbes dplyr et le “pipe” %>%.
Indice pour les ninjas
Notez qu'on peut regrouper un jeu de données par plus d'un facteur, en suivant la syntaxe générale suivante :
group_by(groupe1, groupe2, …)
Cette approche nous permet d'exécuter des opérations sur les sous-groupes contenus dans groupe2 afin the produire un tableau sommaire qui conserve l'information contenu dans groupe1.
dplyr NINJA Défi #6
En utilisant le jeu de données ChickWeight, créez un tableau sommaire nous indiquant, pour chaque Diet, la moyenne de la différence de masse entre la fin et le début de l'étude pour chaque poussin. Utilisez les verbes dplyr et le pipe %>%. (Indice : les fonctions first() et last() pourrait s'avérer utile)
3.5 dplyr - fusion de tableaux de données
En plus de tous les fonctions que nous avons exploré aujourd'hui, dplyr offre également quelques fonctions fort utile nous permettant de réunir 2 tableau de données. La syntaxe de ces fonctions est relativement simple comparé aux autres paquets dans l'arsenal de R :
left_join()right_join()inner_join()anti_join()
Ces fonctions vont au-delà du matériel d'introduction dans cet atelier, mais on vous invite à les explorer et les considérer pour vos propres besoins de manipulation de données.
4. Ressources supplémentaires
Voici quelques ressources intéressantes pour continuer votre apprentissage des paquets ggplot2, tidyr et dplyr. On s'en est d'ailleurs servi comme inspiration pour la matériel couvert aujourd'hui :
ggplot2
Notes du cours Stat 405 de Hadley Wickham (les autres cours sont également géniaux !)
dplyr and tidyr
BONUS ! Allez jeter un coup d'oeil sur le format des scripts recommandé par Google afin de faciliter la compréhension et le partage de ceux-ci :

























