−Table of Contents
AVIS IMPORTANT
Depuis l'automne 2021, ce wiki a été discontinué et n'est plus activement développé.
Tout le matériel mis à jour et les annonces pour la série d'ateliers R du CSBQ se trouvent maintenant sur le site web de la série d'ateliers R du CSBQ. Veuillez mettre à jour vos signets en conséquence afin d'éviter les documents périmés et/ou les liens brisés.
Merci de votre compréhension,
Vos coordonnateurs de la série d’ateliers R du CSBQ.
Ateliers R du CSBQ
Cette série de 10 ateliers guide les participants à travers les étapes requises afin de maîtriser le logiciel R pour une grande variété d’analyses statistiques pertinentes en recherche en biologie et en écologie. Ces ateliers en libre accès ont été créés par des membres du CSBQ à la fois pour les membres du CSBQ et pour la grande communauté d’utilisateurs de R.
Le contenu de cet atelier a été révisé par plusieurs membres du CSBQ. Si vous souhaitez y apporter des modifications, veuillez SVP contacter les coordonnateurs actuels de la série, listés sur la page d'accueil
AVIS IMPORTANT: MISES À JOUR MAJEURES
Mise à jour de mars 2021: Ce wiki n'est plus activement développé ou mis à jour. Le matériel mis à jour pour la série d'ateliers R du CSBQ est maintenant hébergé sur la page GitHub des ateliers R du CSBQ.
Le matériel disponible inclut;
- La présentation Rmarkdown pour cet atelier;
- Un script R qui suit la présentation.
- Le matériel écrit qui accompagne la présentation en format bookdown.
Mise à jour de janvier 2021:
Si vous cherchez l'atelier qui couvre les modèles linéaires généralisés, veuillez vous référer à l'Atelier 6.
Si vous cherchez l'atelier qui couvre les modèles linéaires et généralisés linéaires mixtes, veuillez vous référer à l'Atelier 7.
Atelier 6: Modèles linéaires généralisés
Développé par : Cédric Frenette Dussault, Vincent Fugère, Thomas Lamy, Zofia Taranu
Résumé: Les modèles linéaires généralisés sont des outils importants afin de surmonter un problème récurrent des modèles linéaires, c.-à-d. les variables de réponse n’ayant pas une distribution normale des résidus. Dans cet atelier, vous apprendrez les distributions principales en fonction de la nature de la variable de réponse, le concept de fonction de lien, et comment vérifier les suppositions de base de ces modèles.
Lien vers la nouvelle présentation Rmarkdown
S'il vous plaît essayez-la et dites aux coordonnateurs des ateliers R ce que vous en pensez!
Lien vers l'ancienne présentation Prezi
Téléchargez le script R et les données pour cet atelier :
Limites des modèles linéaires et des modèles linéaires mixtes
Pour illustrer l'utilité des modèles linéaires généralisés (GLMs en anglais), il est important de d'abord comprendre les limites des modèles linéaires (non-généralisés!), soit ceux présentés lors des ateliers 4 et 6. À cet effet, chargeons des données et appliquons quelques modèles linéaires:
- | charger les données
setwd("~/Desktop") mites <- read.csv('mites.csv')
Le jeu de données que vous venez de charger contient une partie du jeu de données des “mites Oribatidae”. Ce dernier a été utilisé par plusieurs ouvrages sur R (p.ex. Borcard, Gillet & Legendre, Numerical Ecology with R) et est disponible dans la bibliothèque “vegan”. Le jeu de données contient 70 échantillons de mousse et de mites provenant de la Station de Biologie des Laurentides de l'Université de Montréal, à Saint-Hippolyte, QC. Chaque échantillon contient des valeurs pour 5 variables environnementales de même que l'abondance de 35 taxa de mites. Le jeu de données que nous utiliserons pour cet atelier contient l'abondance d'un seul taxon de mite, “Galumna sp.”, de même que les valeurs pour les 5 variables environnementales. Notre objectif est de modéliser l'abondance, la présence, et la proportion de Galumna en fonction des 5 variables environnentales. Nous avons donc créé une variable binaire présence/absence (1=présent, 0=absent) et une variable proportion (abondance de Galumna/abondance totale de mites).
- | explorer les données
head(mites) str(mites) # 70 communautés de mites échantillonées à partir de carottes de mousse prises à la Station de Biologie des Laurentides, QC. # Pour chaque carotte/échantillon, les valeurs suivantes sont fournies # $Galumna: abondance de mites du genre Galumna # $pa: présence (1) ou absence (0) de Galumna, peu importe l'abondance # $totalabund: abondance totale de mites, toutes espèces confondues # $prop: proportion de Galumna dans la communauté de mites, i.e. Galumna/totalabund # $SubsDens: densité du substrat # $WatrCont: contenu d'eau de la carotte # $Substrate: type de substrat # $Shrub: abondance de buissons. Traité comme un facteur (une variable qualitative). # $Topo: microtopographie du sol. "couverture" (blanket) ou "hammac" (hummock).
Regardons si nous pouvons voir une relation entre Galumna et les variables environnementales à l'aide d'une simple matrice de diagrammes:
- | matrice de diagrammes
plot(mites)
Il semble y avoir une relation négative entre l'abondance de Galumna et le contenu d'eau (WatrCont). La présence (pa) et la proportion (prop) de Galumna semblent aussi montrer une corrélation négative avec le contenu d'eau. Nous pouvons regarder ça de plus près en représentant dans des diagrammes les relations entre ces 3 variables réponses et avec le contenu d'eau:
- | 3 variables réponses vs. contenu d'eau
par(mfrow=c(1,3)) #division de la fenêtre de diagramme en une ligne et 3 colonnes pour avoir 3 diagrammes sur la même figure. plot(Galumna ~ WatrCont, data = mites, xlab = 'Water content', ylab='Abundance') boxplot(WatrCont ~ pa, data = mites, xlab='Presence/Absence', ylab = 'Water content') plot(prop ~ WatrCont, data = mites, xlab = 'Water content', ylab='Proportion') par(mfrow=c(1,1)) #retour aux paramètres par défaut (un seul diagramme par figure)
En effet, Galumna montre une relation négative avec le contenu d'eau, ce qui suggère que Galumna préfère les sites moins humides. Nous pouvons utiliser des modèles linéaires (fonction “lm”) pour voir si ces relations sont statistiquement significatives.
- | modèles linéaires
lm.abund <- lm(Galumna ~ WatrCont, data = mites) summary(lm.abund) lm.pa <- lm(pa ~ WatrCont, data = mites) summary(lm.pa) lm.prop <- lm(prop ~ WatrCont, data = mites) summary(lm.prop)
Oui, il y a une forte relation significative pour les 3 variables réponses! Ça y est, on soummet à Nature. Attends une minute… Validons d'abord ces modèles pour voir s'ils respectent les suppositions des modèles linéaires, en commençant par le modèle d'abondance.
- | modèle d'abondance
plot(Galumna ~ WatrCont, data = mites) abline(lm.abund)
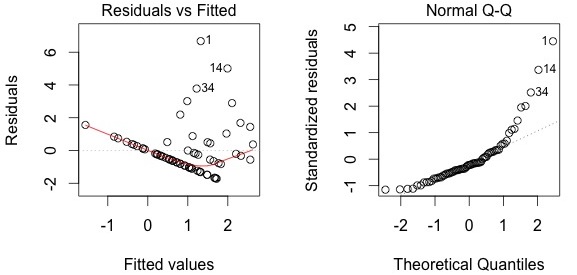
Le modèle ne représente pas très bien les données. Il prédit des valeurs négatives d'abondance lorsque le contenu d'eau excède 600, ce qui est insensé. En plus, le modèle ne prédit pas bien les valeurs élevées d'abondance lorsque le contenu d'eau est faible.
- | diagrammes diagnostiques
plot(lm.abund)
Les diagrammes diagnostiques montrent que le modèle ne respecte pas les suppositions d'homogénéité de la variance (le diagramme à gauche montre que les rédidus sont plus grands pour les valeurs prédites élevées) ni de normalité (le diagramme à droite indique que les résidus ne sont pas distribués tel que prédit par une distribution normale, i.e. plusieurs points sont loin de la ligne pointillée). En conséquence, il nous faut rejeter ce modèle; nous ne pouvons pas l'utiliser pour conclure que l'abondance de Galumna varie en fonction du contenu d'eau. Les diagrammes diagnostiques indiquent que les modèles pour présence-absence et proportion sont eux aussi innapropriés:
- | autres MLs
#Proportion plot(prop ~ WatrCont, data = mites) abline(lm.prop) plot(lm.prop) #Présence/Absence plot(pa ~ WatrCont, data = mites) abline(lm.pa) plot(lm.pa)
C'est très commun que les jeux de données biologiques ne respectent pas les suppositions d'homogénéité de la variance ou de normalité. Ces deux suppositions constituent les problèmes principaux des modèles linéaires, et sont la raison principale pourquoi nous avons besoin des modèles linéaires généralisés. Revoyons l'équation de base d'un modèle linéaire pour mieux comprendre d'où viennent ces suppositions. Cette équation est:
yi = β0 + β1xi + εi, où:
- yi est la valeur prédite pour la variable réponse,
- β0 est l'ordonnée à l'origine de la droite de régression entre y et x,
- β1 est la pente de la droite de régression entre y et x,
- xi est la valeur de la variable indépendante,
- εi sont les résidus du modèle, qui proviennent d'une distribution normale avec une moyenne variable mais une variance constante.
Ce dernier point à propos de εi est important. C'est de là que proviennent les suppositions de normalité et d'homogénéité de la variance (homoscédasticité). Ceci veut dire que les residus du modèle (les distances entre chaque observation et la droite de régression) peuvent être prédits en pigeant de façon aléatoire des valeurs dans une distribution normale. Souvenez-vous que toutes les distributions/lois normales ont deux paramètres, soit μ (la moyenne de la distribution) et σ2 (la variance de la distribution):
Dans un modèle linéaire, μ change selon la valeur de x (la variable indépendante), mais σ2 garde la même valeur pour toutes les valeurs de yi. En effet, une autre équation pour représenter un modèle linéaire est la suivante:
yi ~ N(μ = β0 + β1xi, σ2),
ce qui signifie littéralement que chaque observation (yi) provient d'une distribution normale avec les paramètres μ (qui dépend de la valeur de xi) et σ2. Éssayons de prédire l'abondance de Galumna en fonction du contenu d'eau en utilisant cette équation et les paramètres que nous avons estimé plus tôt avec la fonction lm(). D'abord, il nous faut des valeurs pour β0 et β1 (les coéfficients de régression), que nous pouvons obtenir ainsi:
- | coeffs ML
coef(lm.abund) #(Intercept) WatrCont #3.439348672 -0.006044788
Ce modèle prédit que pour un contenu d'eau de disons 300, nous devrions obtenir une abondance de Galumna de 1.63:
3.44 + (-0.006 x 300) = 1.63.
1.63 est l'abondance prédite s'il n'y avait pas de variance résiduelle (i.e. si notre modèle avait un r2 de 1). Pour modéliser les valeurs prédites, nous devons donc ajouter εi, i.e. la distance entre les valeurs observées et la droite de régression. C'est ici que nous utilisons la distribution normale. Pour x = 300, notre modèle prédit que εi devrait suivre une distribution normale avec une moyenne = 1.63. Lorsque le contenu d'eau = 400, εi devrait suivre une distribution normale avec une moyenne = 3.44 + (-0.006 x 400) = 1.02. Chaque valeur de yi est modélisée en utilisant une distribution normale différente, chacune avec une moyenne distincte qui dépend de xi. Par contre, la variance de toutes ces distributions (σ2) reste toujours la même. La fonction lm() trouve la valeur de σ2 optimale pour minimiser la somme des carrés résiduelle et utilise ensuite cette valeur pour toutes les distributions normales servant à modéliser y. Nous pouvons trouver la valeur de σ2 de notre modèle dans le résumé du modèle:
Nous trouvons que sigma est plus ou moins 1.51. Nous avons maintenant tous les coéfficients nécessaires pour modéliser manuellement l'abondance de Galuman comme le ferait la fonction lm(). À un contenu d'eau de 300, les résidus devraient suivre une distribution normale avec les paramètres μ = 1.63 et σ2 = 1.51. À un contenu d'eau de 400, les résidus devraient suivre une distribution normale avec les paramètres μ = 1.02 et σ2 = 1.51, etc. Graphiquement, ça ressemble à ça:
Les quatres lois normales sur ce graphique donnent la probabilité d'observer toutes valeurs possibles de Galumna à 4 contenus d'eau différents. La moyenne de la distribution varie selon le contenu d'eau (donc μ diminue avec le contenu d'eau), mais σ2 est toujours 1.51. La variance est donc homogène pour toutes les valeurs de x. Ce modèle est inadéquat pour au moins deux raisons:
1. Les valeurs prédites sont en moyenne plus loin de la droite de régression lorsque le contenu d'eau est faible. Ceci signifie que la variance résiduelle est plus grande lorsque x est faible, ce qui revient à dire que εi varie en fonction de x et que la variance n'est donc pas homogène. Il est innaproprié ici d'utiliser une valeur constante de σ2: les distributions normales utilisées pour prédire y devraient être plus larges (avoir une plus grande variance) lorsque x est faible que lorsqu'il est élevé. Malheureusement, les modèles linéaires ne permettent pas ceci.
2. C'est innaproprié d'utiliser une distribution normale pour prédire y en fonction de x. Notre variable réponse est l'abondance qui, par définition, doit être un nombre entier. Pourtant, lorsque le contenu d'eau = 300, notre modèle prédit que l'abondance la plus probable est 1.63! Nous savons pourtant que la probabilité d'observer une telle valeur (ou toute autre fraction) est 0. Nos valeurs prédites devraient être modélisées en utilisant une distribution qui ne contient que des nombres entiers, plutôt qu'avec une distribution continue telle que la distribution normale. Ceci est un problème courant car les variables biologiques peuvent être distribuées de bien d'autres façon que ce qui est prédit par la distribution normale.
Les modèles linéaires généralisés peuvent résoudrent ces deux problèmes. Poursuivez votre lecture!
Les données biologiques et leurs distributions
Les statisticiens ont développé une foule de lois de probabilité (ou distributions) correspondant à divers types de données. Une loi de probabilité donne la probabilité d'observer chaque issu possible d'une expérience ou campagne d'échantillonage (par. ex. abondance = 8 Galumna est un issu d'un échantillonage). Les lois “discrètes” n'incluent que des nombres entiers dans leur ensemble d'issus, alors que les lois “continues” incluent aussi des fractions (par. ex. la loi normale). Toutes les lois ont des paramètres qui déterminent la forme de la loi/distribution (par. ex. μ et σ2 pour la loi normale). Pour un excellent survol des lois de probabilités utiles en écologie, nous vous recommandons le chapitre 4 du livre de Ben Bolker Ecological Models and Data in R. Ici nous ne présenterons que brièvement quelques distributons utiles pour les GLMs.
Nous avons déjà vu que notre variable “abondance de Galumna ” ne peut prendre comme valeur que des nombres entiers, et serait donc mieux modélisée par une loi discrète qu'une loi continue. Une loi utile pour modéliser les données d'abondance est la loi de “Poisson”, nommé en l'honneur du statisticien Siméon Denis Poisson. La loi de Poisson est une loi discrète avec un seul paramètre: λ (lambda), qui détermine et la moyenne et la variance de la distribution (la moyenne et la variance d'une loi de Poisson sont donc égales). Voici 3 exemples de lois de Poisson avec des valeurs différentes de λ, ce qui correspond ici au nombre moyen de Galumna retrouvé dans un ensemble fictif d'échantillons:
Remarquez que lorsque λ est faible (c.-à-d. lorsque la moyenne s'approche de zéro), la distribution est décalée vers la gauche, alors que lorsque λ est élevée, la distribution est symmétrique. La variance augmente aussi avec la moyenne (puisque les deux ont la même valeur), les valeurs prédites sont toujours des nombres entiers, et l'ensemble d'issus d'une loi de Poisson est strictement positif. Toutes ces propriétés sont utiles pour modéliser les données de dénombrement, p.ex. l'abondance d'un taxon, le nombre de graines dans une parcelle, etc. Notre variable mites$Galumna semble suivre une loi de Poisson avec une basse valeur de λ (en effet, si nous calculons l'abondance moyenne de Galumna pour l'ensemble des échantillons avec la fonction mean(), nous observons que cette valeur est proche de 0):
- | histogramme pour abondance de Galumna
hist(mites$Galumna) mean(mites$Galumna)
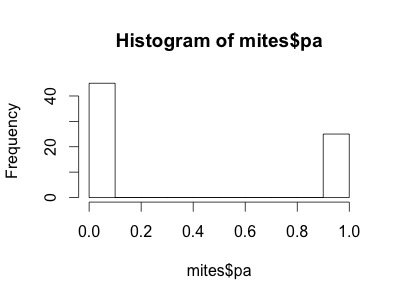
La variable mites$pa (présence-absence) prend une autre forme. Cette variable n'inclue que des 0s et des 1s, de telle sorte que la loi de Poisson ne serait pas plus appropriée pour cette variable que la loi normale.
Nous avons besoin d'une distribution qui n'inclut dans son ensemble que deux issus possibles: 0 ou 1. La loi de “Bernoulli” est une distribution de la sorte. C'est souvent la première loi de probabilité qu'on nous présente dans les cours de statistiques, pour prédire la probabilité d'obtenir “pile” ou “face” en tirant à… pile ou face. Cette loi n'a qu'un paramètre: p, la probabilité de succès. Nous pouvons utiliser la loi de Bernouilli pour calculer la probabilité d'obtenir l'issu “Galumna présent” (1) vs. “Galumna absent” (0). Voici des exemples de distributions de Bernoulli avec différentes probabilités de présence (p):
Nous pouvons calculer le nombre de sites où Galumna est présent par rapport au nombre total de sites pour obtenir un estimé de ce que p pourrait être dans cet exemple:
- | p pour Galumna présence-absence
sum(mites$pa) / nrow(mites)
p pour la variable mites$pa est plus ou moins 0.36, de tel sorte qu'environ deux fois plus de sites montrent l'issu “Galumna absent” (0) que l'issu “Galumna présent” (1).
Lorsqu'il y a plusieurs épreuves (chacune avec un succès/échec), la loi de Bernoulli se transforme en loi binomiale, qui inclue le paramètre additionel n, le nombre d'épeuves. La loi binomiale prédit la probabilité d'observer une certaine proportion de succès, p, sur le nombre total d'épreuves, n. Un “succès” pourrait être, par exemple, la présence d'un taxon à un site (comme pour Galumna), le nombre d'individus qui survit pendant une année, etc. Voici des exemples de lois binomiales avec n = 50 et trois valeurs différentes de p:
Remarquez que la loi binomiale est assymétrique et décalée à gauche lorsque p est faible, mais elle est décalée à droite lorsque p est élevé. C'est la différence principale avec la loi de Poisson: l'étendu de la loi binomial a une limite supérieure, correspondant à n. Conséquemment, la loi binomiale est utilisée pour modéliser des données lorsque le nombre de succès est donné par un nombre entier, et lorsque le nombre d'épreuves est connu. Nous pouvons utiliser la distribution binomiale pour modéliser nos données de proportion, où chaque mite échantillonée pourrait être considérée comme une épreuve. Si la mite est du genre Galumna, l'épreuve est un succès (1), sinon, c'est un échec (0). Dans ce cas, le nombre d'épreuves n varie pour nos 70 échantillons selon l'abondance totale de mites dans l'échantillon, alors que p, la probabilité de succès, est donné par la proportion de Galumna dans chaque échantillon.
Pourquoi tout cette discussion à propos des lois de distribution? Parce que n'importe quelle loi peut être utilisée pour remplacer la loi normale lorsqu'on calcule les valeurs estimées dans un modèle linéaire. Par exemple, nous pouvons utiliser la loi de Poisson pour modéliser nos valeurs d'abondance en utilisant l'équation suivante:
yi ~ Poisson(λ = β0 + β1xi)
Remarquez que λ varie selon x (contenu d'eau), ce qui signifie que la variance dans les résidus variera aussi selon x (car pour la loi de Poisson variance = aussi λ). Ceci veut dire que nous venons de nous défaire de la supposition d'homogénéité de la variance! Aussi, les valeurs estimées seront désormais des nombres entiers plutôt que des nombres décimaux car ils seront tirés de lois de Poisson avec différentes valeurs de λ. Ce modèle ne prédiera jamais de valeurs négatives car l'ensemble d'une loi de Poisson est toujours strictement positif. En changeant la distribution des résidus (εi) de normale à Poisson, nous avons corrigé presque tous les problèmes de notre modèle linéaire pour l'abondance de Galumna.
Ce modèle est presqu'un GLM de Poisson, qui ressemble à ça:
Remarquez que les probabilités d'observer différentes valeurs estimées (en orange) sont maintenant des nombres entiers, et qu'autant la variance que la moyenne de la distribution declinent lorsque λ diminue avec le contenu d'eau. Pourquoi la droite de régression est-elle courbée? Pourquoi est-ce que ce modèle se nomme un “modèle linéaire généralisé”? Continuez votre lecture!
GLM avec une variable réponse binaire
Les variables binaires sont fréquentes en écologie : on observe un phénomène X ou son “absence”. Par exemple, on note souvent la présence ou l'absence d'espèces lors d'études de terrain. Le but est généralement de déterminer si la présence d'une espèce est influencée par différentes variables environnementales. D'autres exemples courants sont la présence/absence d'une maladie au sein d'une population sauvage, l'observation/non-observation d'un comportement spécifique et la survie/mort d'un individu. Un modèle de régression qui utilise une variable binaire comme variable réponse est l'un de plusieurs modèles linéaires généralisés (GLM) et est appelé régression logistique ou modèle logit.
Dans R, la présence (ou succès, survie…) est habituellement codée par un 1 et une absence (ou échec, mort…) par un 0. On effectue une régression logistique (ou n'importe quel GLM) à l'aide de la fonction glm(). Cette fonction diffère un peu de la fonction de base lm(), car elle permet de spécifier une distribution statistique autre que la distribution normale. Nous avons déjà vu que les variables binaires ne sont pas distribuées normalement (i.e. on observe un pic à 0 et un pic à 1 et rien entre les deux). La dernière section a montré que la distribution de Bernoulli est appropriée pour modéliser les variables réponses binaires. La moyenne de cette distribution est représentée par la probabilité p d'observer un résultat et la variance est calculée par p*(1 - p). Le terme (1 - p) représente la probabilité de ne pas observer un résultat. Dans R, on spécifie la distribution statistique du modèle avec l'argument family. Pour la régression logistique, on l'indique de la façon suivante : family = 'binomial'. Rappelez-vous que la distribution de Bernoulli est un cas spécifique de la distribution binomiale lorsque le nombre de répétitions est égal à 1: R “comprend” qu'il faut utiliser une distribution de Bernoulli.
Lorsqu'on prédit la probabilité d'observer un phénomène Y qui est une variable binaire, la valeur prédite doit se trouver entre 0 et 1 : c'est l'étendue possible d'une probabilité ! Si on utilise un modèle linéaire de base pour modéliser une variable binaire en fonction de variables explicatives, il est fort possible qu'on obtienne des valeurs prédites qui se retrouvent en dehors de l'intervalle [0,1], ce qui ne fait aucun sens. L'exemple suivant va vous permettre de mieux comprendre pourquoi un modèle linéaire traditionnel est inapproprié. La prochaine sous-section va vous montrer comment éviter ce problème avec une fonction de lien. Brièvement, une fonction de lien est utilisée pour linéariser la relation entre les valeurs prédites d'un modèle et le prédicteur linéaire (voir sous-section suivante).
- | Utilisation inappropriée d'un modèle linéaire
model.lm <- lm(pa ~ WatrCont + Topo, data = mites) fitted(model.lm) # La fonction "fitted()" extraie les valeurs prédites de la variable réponse du modèle linéaire. # Certaines valeurs sont en-dessous de 0, ce qui ne fait pas de sens pour une régression logistique. # Essayong le même modèle, mais avec une distribution binomiale cette fois-ci. # Remarquez l'argument "family" pour spécifier la distribution. model.glm <- glm(pa ~ WatrCont + Topo, data = mites, family = binomial) fitted(model.glm) # Toutes les valeurs sont comprises entre 0 et 1.
Le concept de la fonction de lien
Afin d'éviter les biais reliés aux modèles linéaires de base, nous avons besoin de spécifier deux choses : une distribution statistique pour les résidus du modèle et une fonction de lien pour les valeurs prédites par ce même modèle. Nous avons déjà vu la distribution de Bernoulli dans la section précédente, alors nous passerons directement à l'explication du rôle de la fonction de lien.
Pour un modèle de régression linéaire d'une variable réponse continue distribuée normalement, l'équation suivante nous permet d'obtenir les valeurs prédites de la variable réponse :
μ = Xβ
où μ est la valeur prédite de la réponse variable, X est la matrice du modèle (i.e. ça représente les variables explicatives) et β correspond aux paramètres estimés à partir des données (i.e. l'intercept et la pente). Le terme Xβ est appelé le prédicteur linéaire. En termes mathématiques, c'est le produit matriciel de la matrice du modèle X et du vecteur des paramètres estimés β. Regardons cela de plus près dans R :
- | Illustration du concept de prédicteur linéaire
# Chargez le jeu de données CO2 utilisé dans un atelier précédent data(CO2) head(CO2) # Construisez un modèle linéaire de l'absorption de CO2 en fonction de la concentration ambiante de CO2. model.CO2 <- lm(uptake ~ conc, data = CO2) # On extraie la matrice du modèle avec la fonction model.matrix(). X <- model.matrix(model.CO2) # Les paramètres estimés sont extraits ainsi : B <- model.CO2$coefficients # On multiple X et B pour obtenir le prédicteur linéaire. # Le symbole "%*%" indique qu'on veut effectuer le produit matriciel. XB <- X %*% B # On compare les valeurs de XB aux valeurs obtenues avec la fonction fitted(). # Toutes les déclarations devraient être vraies (i.e. TRUE). # On utilise la fonction round() pour que tous les éléments aient cinq décimales. round(fitted(model.CO2), digits = 5) == round(XB, digits = 5)
Lorsqu'on crée un modèle linéaire simple avec une variable réponse continue distribuée normalement, le prédicteur linéaire est égal aux valeurs attendues de la variable réponse. Ceci n'est pas exact si la variable réponse n'est pas distribuée normalement. Si c'est le cas, il faut appliquer une transformation sur les valeurs prédites, i.e. une fonction de lien. La fonction de lien peut être vue comme une transformation des valeurs prédites pour obtenir une relation linéaire entre celles-ci et le prédicteur linéaire :
g(μ) = Xβ
où g(μ) est la fonction de lien des valeurs prédites. Ceci permet d'enlever la contrainte de distribution normale des résidus. Lorsque la variable réponse est une variable binaire, la fonction de lien est la fonction logit et est représentée par l'équation suivante :
logit(μ) = log (μ / 1-μ) = Xβ
où μ représente les valeurs prédites (i.e. la probabilité que Y = 1, car on observe la présence d'une espèce, d'une maladie, d'un succès ou d'un autre événement). Le ratio μ / 1-μ représente la cote (odds en anglais) qu'un événement se produise. Cela transforme les valeurs prédite sur une échelle de 0 à l'infini. Par exemple, si on a une probabilité de 0,8 d'observer une espèce X, la cote est donc de 4 : il est 4 fois plus probable d'observer l'espèce que de ne pas l'observer - 0.8/(1-0.8) = 4. La transformation log (on appelle maintenant ce ratio le log odds) permet aux valeurs d'être distribuées de moins l'infini à l'infini. Donc, la fonction de lien logit prend les valeurs prédites du modèle et les transforme en une variable continue sans borne. Les valeurs prédites peuvent ainsi être reliées à un prédicteur linéaire. C'est pour cette raison qu'on appelle ce modèle un modèle linéaire généralisé même si la relation entre la variable réponse et la variable explicative ne ressemble pas nécessairement à une “ligne droite” !
- | Un exemple simple de régression logistique
# On construit un modèle de régression de la présence/absence d'une espèce de mite (Galumna sp.) # en fonction du contenu en eau du sol et de la topographie. # On utilise la fonction glm() et on spécifie l'argument "family". logit.reg <- glm(pa ~ WatrCont + Topo, data = mites, family = binomial(link = "logit")) # La fonction de lien "logit" est la fonction par défaut pour une régression logistique, # ce qui signifie qu'il n'est pas nécessaire de l'indiquer avec l'argument "family": logit.reg <- glm(pa ~ WatrCont + Topo, data = mites, family = binomial) summary(logit.reg)
DÉFI 1
En utilisant le jeu de données bacteria (du paquet MASS), modélisez la présence de H. influenzae en fonction du traitement (trt) et de la semaine de test (week). Commencez avec un modèle saturé et réduisez-le jusqu'à obtenir le modèle le plus parcimonieux possible.
Interpréter la sortie d'une régression logistique
La sortie du modèle de régression logistique indique que les deux variables explicatives (WatrCont et Topo) sont significatives, mais comment interprète-on les coefficients estimés ? Rappelez-vous que nous avons effectué une transformation des valeurs prédites par le modèle (i.e. la probabilité que Y = 1), alors il faut utiliser une fonction inverse pour pouvoir interpréter correctement les résultats. On peut utiliser la fonction exponentielle ex pour obtenir la cote de probabilité pour chaque variable explicative.
- | Interpréter la sortie d'une régression logistique
# Pour obtenir les pentes, il faut utiliser la fonction exponentielle exp(). # Ceci mettra les coefficients sur l'échelle des cotes. # Mathématiquement, ceci correspond à : # exp(model coefficients) = exp(log(μ / (1 - μ)) = u / (1 - μ) # C'est notre rapport de cotes ! exp(logit.reg$coefficients[2:3]) # WatrCont TopoHummock # 0.9843118 8.0910340 # Pour obtenir l'intervalle de confiance sur l'échelle des cotes : exp(confint(logit.reg)[2:3,]) # 2.5 % 97.5 % # WatrCont 0.9741887 0.9919435 # TopoHummock 2.0460547 38.6419693
Prenez note que la cote pour une variable explicative est calculée lorsque les autres variables sont gardées constantes. La topographie a une cote de 8.09. Ceci signifie que la probabilité d'observer Galumna sp. est 8.09 fois plus vraisemblable lorsque la topographie est de type hummock plutôt que blanket.
Lorsque la cote est inférieure à 1, l'interprétation est un peu plus compliquée. Si c'est le case, il faut prendre la valeur inverse de la cote (i.e. 1 divisé par la cote) pour faciliter l'interprétation. L'interprétation revient à dire comment l'observation d'un phénomène est MOINS probable. Pour le contenu en eau du sol, la cote est de 0.984. L'inverse est 1 / 0.984 = 1.0159. Ceci signifie que l'augmentation d'une unité en contenu en eau diminue la vraisemblance d'observer la présence de Galumna sp. de 1.0159. On peut aussi l'exprimer en pourcentage en soustrayant 1 à cette valeur : (1.0159 - 1) * 100 = 1.59 %. Il est 1.59 % moins vraisemblable d'observer Galumna sp. avec une augmentation d'une unité de contenu en eau. Pour se convaincre qu'on fait la bonne interprétation, on peut représenter graphiquement les résultats de la présence de Galumna sp. en fonction du contenu en eau du sol. On voit qu'en moyenne la présence de Galumna sp. est plus élevée lorsque le contenu en eau est faible
Lorsqu'un paramètre estimé est entre 0 et 1 sur l'échelle des cotes, la relation entre la variable réponse et la variable explicative est négative. Si la valeur est supérieure à 1, cela indique une relation positive entre les variables. Si l'intervalle de confiance inclut la valeur 1, la relation entre les variables n'est pas significative. Rappelez-vous qu'une valeur de 1 sur l'échelle des cotes signifie que la probabilité d'observer un phénomène Y est la même que celle de ne pas observer ce phénomène (i.e. quand p = 0.5, 0.5/(1-0.5) = 1).
Pour obtenir une probabilité au lieu d'une cote pour chaque variable explicative, il faut utiliser la fonction logit inverse :
logit-1 = 1/(1+1/exp(x))
où x est le paramètre à transformer de l'échelle log odds à l'échelle de probabilité. Pour le modèle logit.reg, le paramètre estimé pour la topographie est de 2.091 sur l'échelle log odds. Donc, la probabilité est donnée par :
1/(1+1/exp(2.091)) = 0.89 ce qui équivaut à 1/(1+1/8.09). Rappelez-vous que la valeur 8.09 est sur l'échelle des cotes. On a une probabilité de 0.89 d'observer Galumna sp. lorsque la topographie est de type hummock.
- | Un peu de mathématique sur la fonction logit inverse
# On commence avec la valeur de cote pour la topographie du modèle logit.reg : µ/ (1 - µ) = 8.09 # On réarrange pour isoler µ : µ = 8.09(1 - µ) = 8.09 - 8.09µ 8.09µ + µ = 8.09 µ(8.09 + 1) = 8.09 µ = 8.09 / (8.09 + 1) µ = 1 / (1 + (1 / 8.09)) = 0.89 # On obtient le même résultat sans utiliser la fonction logit inverse !
Pouvoir prédictif et validation du modèle
Une façon simple et intuitive d'estimer le pouvoir explicatif d'un GLM est de comparer la déviance du modèle à celle d'un modèle nul. La déviance peut être vue comme une généralisation du concept de la somme des carrés résiduelle lorsque le modèle est estimé par maximisation de la vraisemblance (i.e. la méthode par laquelle on estime les paramètres d'un GLM). Ceci nous permet de calculer un pseudo-R2, une statistique similaire au R2 dans une régression des moindres carrés (i.e. la méthode utilisée pour estimer les paramètres d'une régression linéaire de base). Le modèle nul correspond à un modèle sans variable explicative. Dans R, on l'indique de la façon suivante : null.model <- glm(Response.variable ~ 1, family = binomial). La forme générique pour calculer un pseudo-R2 est :
Pseudo-R2 = (déviance du modèle nul – déviance résiduelle) / déviance résiduelle
où “déviance du modèle nul” est la déviance du modèle nul et “déviance résiduelle” est la déviance résiduelle du modèle d'intérêt. La différence est divisée par la déviance du modèle nul afin de contraindre le pseudo-R2 entre 0 et 1.
- | Le pseudo-R2 d'une régression logistique
# Les déviances résiduelle et nulle sont déjà enregistrées dans un objet de type glm. objects(logit.reg) pseudoR2 <- (logit.reg$null.deviance – logit.reg$deviance) / logit.reg$null.deviance pseudoR2 # [1] 0.4655937
Les variables explicatives du modèle expliquent 46.6% de la variabilité de la variable réponse.
Récemment, Tjur (2009) a proposé une nouvelle statistique, le coefficient de discrimination (D), afin d'évaluer le pouvoir prédictif d'une régression logistique. Intuitivement, D évalue si la régression logistique peut classer adéquatement chaque résultat comme un succès ou un échec. Mathématiquement, c'est la différence entre la moyenne des valeurs prédites des succès (i.e. Y = 1) et des échecs (i.e. Y = 0) :
où est la moyenne attendue des probabilités lorsqu'un événement est observé et
est la moyenne attendue des probabilités lorsqu'un événement n'est pas observé. Une valeur de D près de 1 indique que le modèle accorde une probabilité élevée d'observer un événement lorsque celui-ci a été observé et une probabilité faible d'observer un événement lorsque celui-ci n'a pas été observé. Une valeur de D près de 0 indique que le modèle n'est pas utile pour distinguer entre les observations et les “non-observations” d'un résultat. Le code suivant montre comment obtenir la valeur de D et comment représenter visuellement les valeurs de
et
.
- | Le coefficient de discrimination et sa visualisation
install.packages("binomTools") library("binomTools") # La fonctions Rsq calcule indices d'ajustement # dont le coefficient de discrimination. # Pour plus d'information sur les autres indices, consultez Tjur (2009). # Les histogrammes montrent la distribution des valeurs attendues lorsque le résultat est observé # et non observé. # Idéalement, le recouvrement entre les deux histogrammes doit être minimal. fit <- Rsq(object = logit.reg) fit # R-square measures and the coefficient of discrimination, 'D': # # R2mod R2res R2cor D # 0.5205221 0.5024101 0.5025676 0.5114661 # # Number of binomial observations: 70 # Number of binary observation: 70 # Average group size: 1 plot(fit, which = "hist")
Pour évaluer l'ajustement (i.e. goodness-of-fit) d'une régression logistique, les graphiques de diagnostic ne sont pas très utiles (voir atelier 4). On utilise plutôt un test de Hosmer-Lemeshow pour évaluer si un modèle est approprié ou non. Ce test sépare les valeurs attendues (ordonnées de la plus faible à la plus grande) en groupes de même taille. Dix groupes est généralement le nombre de groupes recommandé. Pour chaque groupe, on compare les valeurs observées aux valeurs attendues. Le test est similaire à un test de khi-carré avec G - 2 degrés de liberté (G est le nombre de groupes). Dans R, ce test est disponible dans le paquet binomTools ou vcdExtra.
- | Le test de Hosmer-Lemeshow
fit <- Rsq(object = logit.reg) HLtest(object = fit) # La valeur de p est de 0.9051814. Donc, on ne rejète pas notre modèle. # L'ajustement du modèle est bon.
DÉFI 2
Évaluez l'ajustement et le pouvoir prédictif du modèle model.bact2.
Comment pouvez-vous améliorer le pouvoir prédictif du modèle ?
Représentation graphique des résultats
Lorsque le modèle a été validé, il peut être utile de représenter les résultats graphiquement afin de voir comment la variable est influencée par les variables explicatives. Une façon de faire est de mettre à la fois les valeurs observées et prédites de la variable réponses en fonction d'une variable explicative sur un même graphique. Voici un exemple avec le paquet ggplot2. Revoir l'atelier 3 pour plus d'informations sur ce paquet.
- | Représenter graphiquement les résultats d'une régression logistique
library(ggplot2) ggplot(mites, aes(x = WatrCont, y = pa)) + geom_point() + stat_smooth(method = "glm", family= "binomial", se = FALSE) + xlab("Water content") + ylab("Probability of presence") + ggtitle("Probability of presence of Galumna sp. as a function of water content")
Un exemple avec des données de proportions
Les données de proportions sont plus près des variables binaires que vous ne l'imaginez ! Supposons que vous êtes sur le terrain pour estimer la prévalence d'une maladiechez le cerf de Virginie le long d'un gradient environnemental de disponibilité en ressources. Vous échantillonnez dix individus dans dix populations différentes pour estimer la prévalence de la maladie. Votre feuille de données comporte dix lignes avec l'information suivante : code de la population et nombre d'individus infectés dans la population. À première vue, on dirait des données d'occurrences (i.e. count data), mais ce n'est pas correct de le voir ainsi : vous savez combien d'individus vous avez échantillonnés dans chaque population. Vous avez donc des données de proportions : c'est le nombre d'individus infectés sur dix individus échantillonnés. Si vous vous rappelez ce que vous avez lu dans la section sur les distributions, vous devez choisir une distribution binomiale pour modéliser ces données. Illustrons ceci avec un exemple :
- | Un GLM avec des données de proportions
# Commençons par générer des données en se basant sur l'exemple précédent : # On choisit un nombre aléatoire entre 1 et 10 pour déterminer le nombre de cerfs infectés (objet 'n.infected'). # On échantillonne dix individus dans dix populations différentes (objet 'n.total'). # 'res.avail' est un indice de qualité de l'habitat (disponibilité de ressources). set.seed(123) n.infected <- sample(x = 1:10, size = 10, replace = TRUE) n.total <- rep(x = 10, times = 10) res.avail <- rnorm(n = 10, mean = 10, sd = 1) # Ensuite, on spécifie le modèle. Prenez note comment on spécifie la variable réponse. # Il faut indiquer le nombre de fois où la maladie a été détectée # et le nombre de fois où celle-ci n'a pas été détectée. prop.reg <- glm(cbind(n.infected, n.total - n.infected) ~ res.avail, family = binomial) summary(prop.reg) # Si vos données sont déjà sous la forme de proportions, voici comment faire dans R : # Créons tout d'abord un vecteur de proportions : prop.infected <- n.infected / n.total # On doit spécifier l'argument "weights" de la fonction glm() pour indiquer le nombre d'essais par site. prop.reg2 <- glm(prop.infected ~ res.avail, family = binomial, weights = n.total) summary(prop.reg2) # Les sommaires des modèles prop.reg et prop.reg2 sont identiques !
GLM avec des données d'abondance
Afin d'illustrer l'utilisation des GLMs avec des données d'abondance, nous allons utiliser un nouveau jeux de données; faramea.
- | loading data
faramea <- read.csv(‘faramea.csv’, header = TRUE)
Ce jeux de données s'intéresse à l'espèce d'arbre Faramea occidentalis sur l'île Barro Colorado au Panama. 43 transects ont été utilisés afin de mesurer le nombre d'arbre le long d'un gradient environnemental. Des caractéristiques environnementales, comme l'élévation du terrain et la précipitation, ont aussi été mesurées au niveau de chaque transect. Examinons maintenant à quoi ressemble la distribution du nombre d'arbres par transect.
- | plot the data
hist(faramea$Faramea.occidentalis, breaks=seq(0,45,1), xlab=expression(paste("Number of ", italic(Faramea~occidentalis))), ylab="Frequency", main="", col="grey")
Nous pouvons remarquer qu'il n'y a que des valeurs entières et positives. Etant donné cette spécificité propore aux données de dénombrement, la distribution de Poisson, décrite ci dessus, semble un choix approprié pour modéliser ces données.
GLM avec une distribution de Poisson
Comme nous l'avons vu plus haut, la distribution de Poisson est particulièrement appropriée pour modéliser des données de dénombrement car :
- elle ne spécifie des probabilités que pour des valeurs entières
- P(y<0) = 0, en d'autres termes la probabilité d'observer une valeur négative est nulle
- la relation entre la moyenne et la variance permet de manipuler des données hétérogènes (e.g. quand la variance dans les données augmente avec la moyenne)
Dans notre exemple, nous voulons tester si l'élévation (une variable explicative continue) influence l'abondance de Faramea occidentalis. Il est généralement bien de commencer par utiliser un GLM et une distribution de Poisson lorsque nous cherchons à modéliser des données d'abondance. Nous allons donc commencer par ajuster ce type de modèle pour modéliser l'abondance de Faramea occidentalis en fonction de l'élévation.
Mais qu'est ce qui se cache derrière un tel GLM ?
un GLM avec une distribution de Poisson considère que les valeurs yi de la variable réponse ont été générées par une distribution de Poisson dont la moyenne et la variance sont égales à µi.
Yi ∼ Poisson(µi)
Avec E(Yi) = Var(Yi) = µi
Souvenez vous qu'un GLM est composé de deux parties, le prédicteur linéaire et la fonction de lien. Le prédicteur linéaire est utilisé comme une combinaison linéaire des différentes variables explicatives dont les effets sont estimés à travers les paramètres β. Le prédicteur linéaire peut être défini comme :
β0 + Xi.β
où X est la matrice des variables explicatives, β0 l'ordonnée à l'origine et β le vecteur des paramètres estimés.
La fonction de lien, entre le prédicteur linéaire et la moyenne de la distribution µi est la fonction logarithmique (relation log-linéaire) :
- log(µi) = β0 + Xi.β
ou
- µi = exp(β0 + Xi.β)
La distribution de Poisson donne la probabilité qu'une valeur Yi soit observée étant donné une moyenne µi = exp(β0 + Xi.β). Dans ce modèle, les paramètres inconnus sont inclus dans le vecteur des coefficients de régression β (plus l'ordonnée à l'origine β0) et seront estimés à l'aide du maximum de vraisemblance.
Pour ajuster un GLM avec une distribution de Poisson sous R, il suffit de spécifier family = poisson dans la fonction glm(). Par défaut la fonction de lien est la fonction logarithmique.
- | fit a Poisson GLM
glm.poisson = glm(Faramea.occidentalis~Elevation, data=faramea, family=poisson) summary(glm.poisson)
Le résumé est similaire à celui de la fonction 'lm' (voir atelier 4) et donne les estimations des paramètres. Vous pouvez aussi récupérer les estimations des paramètres à l'aide des fonctions suivantes :
- | parameter estimates
# ordonnée à l'origine summary(glm.poisson)$coefficients[1,1] # coefficient de regression de l'élévation summary(glm.poisson)$coefficients[2,1]
La validation du modèle et le problème de la surdispersion
Un aspect important du résumé se trouve dans les dernières lignes :
- | Poisson GLM summary
summary(glm.poisson) # Null deviance: 414.81 on 42 degrees of freedom # Residual deviance: 388.12 on 41 degrees of freedom
L'estimation du maximum de vraisemblance est utilisé afin d'estimer les paramètres. Nous avons déjà mentionné que la déviance est l'équivalent en maximum de vraisemblance des sommes des carrés dans un modèle linéaire. Ici vous pouvez considérer la déviance nulle et la déviance résiduelle comme les équivalents de la somme totale des carrés et de la somme des carrés résiduelle. La déviance résiduelle correspond à deux fois la différence entre la log-vraisemblance de deux modèles : un modèle qui s'ajuste parfaitement aux données (i.e. un modèle saturé) et le modèle que nous voulons tester. Si notre modèle est correct, la distribution de la déviance résiduelle est estimée selon une distribution du χ² avec n-p-1 degrés de liberté (où n correspond au nombre d'observations et p correspond au nombre de variables explicatives). Une implication très importante en ce qui nous concerne est que la déviance résiduelle doit être égale au nombre de degrés de liberté résiduels. Dans notre example, la déviance résiduelle vaut 388.12, tandis que nous avons 41 (43-1-1) degrés de liberté. La déviance est 9.5 fois supérieure au nombre de dégrés de liberté. Le modèle peut alors être qualifié de surdispersé.
La surdispersion La surdispersion peut être évaluée à l'aide du paramètre de surdispersion φ qui se mesure donc de la facon suivante :
φ = déviance résiduelle / dégrés de liberté résiduels * φ < 1 indique qu'il y a sousdispersion * φ = 1 indique que la dispersion est conforme aux attendus * φ > 1 indicates qu'il y a surdispersion
Mais pourquoi un GLM présente-il de la surdispersion ? En fait, des modèles GLM sont surdispersés quand la variance dans les données est encore plus grande que ce qu'autorise la distribution de Poisson. Par exemple, cela peut se produire lorsque les données contiennent de nombreux zeros ou beaucoup de très grosses valeurs. Si nous revenons sur la distribution de nos données (ci dessus) nous pouvons remarquer que ces deux problèmes sont présents et que la distribution de Poisson n'était peut être pas le choix idéal. La surdispersion peut aussi survenir lorsque des variables explicatives ou des termes d'intéractions sont absentes ou bien encore lorsque qu'il y a des problèmes de valeurs aberrantes.
La distribution de Poisson peut tenir compte de l'hétérogénéité présente dans des données grace à la relation entre sa moyenne et sa variance. Toutefois dans certains cas la variance augmente bien plus rapidement par rapport à la moyenne si bien que la distribution de Poisson n'est plus appropriée. Pour nous convaincre une dernière fois d'abandonner la distribution de Poisson pour modéliser l'abondance de l'espèce faramea nous pouvons rapidement calculer la moyenne et la variance dans notre jeux de données :
- | mean and variance of the data
mean(faramea$Faramea.occidentalis) var(faramea$Faramea.occidentalis)
Dans la pratique, les GLMs basés sur la distribution de Poisson sont très pratique pour décrire la moyenne µi mais vont sous-estimer la variance dans les données dès qu'il y a de la surdispersion. Par conséquent, les tests qui découlent du modèle seront trop laxistes. Il y a deux moyens de traiter les problèmes de surdispersion que nous allons détailler ci-dessous :
- corriger la surdispersion en utilisant un GLM quasi-Poisson
- choisir une nouvelle distribution comme la binomiale négative
GLM avec une correction "quasi" Poisson
Le principe d'un GLM avec une correction “quasi” Poisson est très simple; le paramètre de surdispersion (φ) est ajouté dans l'équation qui spécifie la variance du modèle :
E(Yi) = µi
Var(Yi) = φ.µi
Le prédicteur linéaire, ainsi que la fonction de lien (log) restent les mêmes. La seule différence est que φ va être estimé afin de corriger le modèle. Les estimations des paramètres seront eux aussi inchangés, mais leurs écarts-types seront multiplés par √φ. Ainsi, certains paramètres qui étaient marginalement significatifs peuvent ne plus le rester.
Dans R, la famille 'quasipoisson' peut être utilisée pour traiter ces problèmes de surdispersion (de la même manière la famille quasibinomial' peut être utilisée). L'estimation de φ sera donné dans le résumé du modèle GLM quasi Poisson. Nous pouvons ajuster ce modèle de deux manières différentes :
- | fit a new quasi-Poisson GLM
# Option 1, nous ajustons un nouveau modèle GLM quasi-Poisson glm.quasipoisson = glm(Faramea.occidentalis~Elevation, data=faramea, family=quasipoisson) # Option 2, nous actualisons le modèle précédent : glm.quasipoisson = update(glm.poisson,family=quasipoisson) # regardons le résumé summary(glm.quasipoisson)
En examinant le résumé du modèle nous pouvons voir que φ est estimé à 15.97. Nous avons donc eu raison de corriger le modèle afin de prendre en compte la surdispersion. Par contre, si nous regardons la significativité du coefficient de regression associé à l'élévation, nous remarquons qu'il n'est plus significatif. Cependant, 15.97 indique que la surdispersion est forte et en général un GLM quasi-Poisson est favorisé lorsque φ est compris entre 1 et 15. Dans un soucis pédagogique, nous allons considérer que nous ne sommes pas satisfait avec ce modèle et que nous voulons maintenant ajuster une distribution binomiale négative à nos données.
Deux points sont importants à garder en tête lorsque vous utilisez un GLM quasi-Poisson afin de corriger la surdispersion :
- Les GLMs quasi-Poisson n'ont pas d'AIC. En effet, la vraisemblance d'un modèle GLM quasi-Poisson ne peut pas être spécifiée et s'appuie sur une procédure de pseudo-maximum de vraisemblance. Par conséquence les GLMs quasi-Poisson n'ont pas d'AIC, et ce critère ne peut pas être utilisé afin de comparer différents modèles. Toutefois des alternatives ont été developpées pour gérer cette situation (e.g. quasi-AIC).
- La surdispersion influence la comparaison de modèles. En effet, la surdispersion influence la comparaison de deux modèles emboités et doit donc être prise en considération. Par exemple, considérons que nous voulons comparer le modèle GLM1, qui contient p1 paramètres avec le modèle GLM2, qui contient p2 paramètres. GLM1 est emboité dans GLM2 et p2 > p1. La comparaison des deux modèles est basées sur le test du rapport des vraisemblances des deux modèles, D1 et D2 respectivement. Si la surdispersion est connue, les déviances doivent être corrigées de manière approprié selon D* = D/φ, et le test final sera basé sur le critère D1* - D*2 qui est supposé être distributé selon une distribution du χ² avec p2-p1 degrés de liberté lorsque le modèle GLM1 est correct.
- Mais dans certain cas φ n'est pas connu. Par exemple, lorsque vous spécifiez un GLM avec un distribution normale. Dans ce cas, φ peut être estimé a posteriori en utilisant la déviance résiduelle du plus gros modèle de telle sorte que le critière de comparaison devienne [(D1-D2)/(p2-p1)]/[D2/(n-p2)]. Ce critère est supposé suivre une distribution F avec p2-p1 et n-p2 degrés de liberté.
GLM avec une distribution binomiale négative
Un GLM avec une distribution binomiale négative (BN) est utilisé lorsque la surdispersion est très forte. La distribution BN contient un paramètre supplémentaire, k, qui va être très utile pour gérer les problèmes de surdispersion. Avant de rentrer dans les détails sur R, voyons rapidement ce qui se cache derrière la distribution BN. En fait, la distribution BN est la combinaison de deux distributions; une distribution de Poisson et une distribution Gamma. La distribution BN définie la distribution d'une variable aléatoire discrète de la même manière qu'une distribution de Poisson mais autorise la variance à être différente de la moyenne. Le mélange entre la distribution de Poisson et la distribution Gamma peut se résumer à l'aide de deux paramètres, µ et k qui spécifie la distribution de la facon suivante :
Y ~ BN(µ, k)
E(Y) = µ et Var(Y) = µ + µ²/k
De cette manière nous pouvons voir comment cette distribution va gérer la surdispersion dans les modèles GLM. Le deuxième terme de la variance de la distribution BN va déterminer le degré de surdispersion. En effet, la surdispersion est indirectement déterminée par k, que représente le paramètre de dispersion. Si k est grand (par rapport à μ²), la deuxième partie de la variance, µ²/k va s'approcher de 0, et la variance de Y sera μ. Dans ce cas la distribution BN converge vers la distribution de Poisson et vous pourriez tout aussi bien utiliser cette dernière. Par contre, plus k sera petit et plus la surdispersion sera grande. Comme avec toutes les autres distributions, un GLM avec une distribution BN se spécifie en trois étapes. Tout d'abord le modèle fait l'hypothèse que les Yi suivent une distribution BN de moyenne μi et de paramètre k.
Yi ∼ BN(µi, k)
E(Yi) = µi et Var(Yi) = µi + µi²/k
Les deux dernières étapes définissent le prédicteur linéaire ainsi que la fonction de lien entre la moyenne des Yi et le prédicteur linéaire. La fonction de lien utilisée par les GLMs avec une distribution BN est le logarithme ce qui permet de s'assurer que les valeurs prédites soient toujours positives.
- log(µi) = β0 + Xi.β
ou
- µi = exp(β0 + Xi.β)
La distribution NB n'est pas dans la fonction glm(), il faut donc installer et charger la librairie MASS. Si vous ne vous souvenez plus si cette librairie R est déjà installée, pas de problème utilisez la fonction suivante :
- | 'Mass' est il installé?
ifelse(length(which(installed.packages() == "MASS")) == 0, {print("MASS not installed. Installing... "); install.packages("MASS")}, print("MASS already installed"))
Sinon, si vous savez que cette librairie n'est pas installée, vous pouvez directement taper la commande :
- | Installer 'MASS'
install.packages("MASS")
Souvenez vous de charger la librairie
- | Charger 'MASS'
library("MASS")
Vous pouvez ajuster un GLM avec une distribution BN à l'aide de la fonction glm.nb() :
- | Ajuster un GLM avec un BN'
glm.negbin = glm.nb(Faramea.occidentalis~Elevation, data=faramea) summary(glm.negbin)
Le résumé du modèle et similaire à celui des autres GLMs (e.g. GLMs Poisson). Cependant vous avez maintenant un nouveau paramètre, theta, qui est le paramètre k de la variance de votre distribution. L'écart-type de ce paramètre est aussi fourni, mais attention à son interprétation car l'intervalle n'est pas symétrique.
Représentation graphique du modèle final
Le GLM avec une distribution BN semble être le meilleur modèle pour modéliser nos données. Nous voulons maintenant représenter la relation entre le nombre de Faramea occidentalis et l'élévation.
- | Représenter le GLM'
# représenter les données observées plot(faramea$Elevation, faramea$Faramea.occidentalis, xlab="Elevation (m)", ylab=expression(paste("Number of", " ", italic(Faramea~occidentalis))), pch=16, col=rgb(4,139,154,150,maxColorValue=255)) # récupérons les valeurs de l'ordonnée à l'origine et de beta à partir du résumé et ajoutons les dans l'équation du modèle curve(exp(summary(glm.negbin)$coefficients[1,1]+summary(glm.negbin)$coefficients[2,1]*x),from=range(faramea$Elevation)[1],to=range(faramea$Elevation)[2],add=T, lwd=2, col="orangered") # récupérons les écarts-types pour construire l'intervalle de confiance du modèle curve(exp(summary(glm.negbin)$coefficients[1,1]+1.96*summary(glm.negbin)$coefficients[1,2]+summary(glm.negbin)$coefficients[2,1]*x+1.96*summary(glm.negbin)$coefficients[2,2]),from=range(faramea$Elevation)[1],to=range(faramea$Elevation)[2],add=T,lty=2, col="orangered") curve(exp(summary(glm.negbin)$coefficients[1,1]-1.96*summary(glm.negbin)$coefficients[1,2]+summary(glm.negbin)$coefficients[2,1]*x-1.96*summary(glm.negbin)$coefficients[2,2]),from=range(faramea$Elevation)[1],to=range(faramea$Elevation)[2],add=T,lty=2, col="orangered")
Nous pouvons voir que le nombre de Faramea occidentalis diminue de manière significative avec l'élévation. Toutefois, l'intervalle de confiance autour de notre modèle est assez large, notamment pour à faible élévation.
Conclusion sur les GLM avec des données d'abondance
Tous les GLMs que nous venons de voir pour modéliser des données de d'abondance (Poisson, quasi-Poisson et BN) utilisent la même relation log-linéaire entre moyenne et prédicteur linéaire (log(µ) = X.β). Toutefois ils vont autoriser différentes relations entre la moyenne et la variance et vont aussi se reposer sur des méthodes d'estimation de la vraisemblance différentes. Les GLMs Quasi-Poisson ou BN sont privilégiés afin de traiter la surdispersion. Malheureusement dans certains cas les données peuvent contenir trop de zeros et d'autres modèles seront plus éfficaces pour traiter ces situations. C'est par exemple le cas des “zero-augmented models” (e.g. zero-inflated Poisson [ZIP]) qui vont traiter les zéros indépendamment des autres valeurs.