Table of Contents
AVIS IMPORTANT
Depuis l'automne 2021, ce wiki a été discontinué et n'est plus activement développé.
Tout le matériel mis à jour et les annonces pour la série d'ateliers R du CSBQ se trouvent maintenant sur le site web de la série d'ateliers R du CSBQ. Veuillez mettre à jour vos signets en conséquence afin d'éviter les documents périmés et/ou les liens brisés.
Merci de votre compréhension,
Vos coordonnateurs de la série d’ateliers R du CSBQ.
Série d'ateliers en R du CSBQ
Cette série de 10 ateliers guide les participants à travers les étapes requises afin de maîtriser le logiciel R pour une grande variété d’analyses statistiques pertinentes en recherche en biologie et en écologie. Ces ateliers en libre accès ont été créés par des membres du CSBQ à la fois pour les membres du CSBQ et pour la grande communauté d’utilisateurs de R.
Le contenu de cet atelier a été révisé par plusieurs membres du CSBQ. Si vous souhaitez y apporter des modifications, veuillez SVP contacter les coordonnateurs actuels de la série, listés sur la page d'accueil
Atelier 5: Programmation en R
Développé par: Johanna Bradie, Sylvain Christin, Ben Haller, Guillaume Larocque
Résumé: Cet atelier vise à vous apprendre les bases de la programmation en R. Vous apprendrez à utiliser des structures de contrôle (boucles for, if, while) afin d'éviter la répétition de code, de faciliter l'organisation et d'effectuer des simulations. Vous apprendrez également à écrire vos propres fonctions et quelques astuces pour programmer plus efficacement. La dernière partie de l'atelier portera sur des librairies de R qui peuvent être très utiles pour les participants, mais qui ne seront pas couvertes ailleurs dans la série d'ateliers en R du CSBQ.
Lien vers la nouvelle présentation Rmarkdown
S'il vous plaît essayez-la et dites aux coordonnateurs des ateliers R ce que vous en pensez!
Lien vers l'ancienne présentation Prezi
Téléchargez le script R pour cet atelier:
Objectifs d'apprentissage
- Structures de contrôle
- Écriture de fonctions en R
- Réduire le temps d’exécution des codes
- Paquets R utiles pour les biologistes
Contrôle de flux
En programmation, le contrôle de flux est simplement l'ordre dans lequel le programme est exécuté.
Pourquoi est-il avantageux de structurer nos programmes?
- Réduit la complexité et la durée de la tâche en question
- Une structure logique améliore la clarté du code
- Plusieurs programmeurs peuvent aussi travailler sur un même programme
Tout ceci augmente la productivité
On peut utiliser des organigrammes pour planifier nos programmes et leur structure.
Représenter la structure
Les deux composantes de base de programmation sont:
La sélection
Exécuter des commandes conditionnellement en utilisant:
if if else
L'itération
Répéter l'exécution d'une commande tant qu'une condition n'est pas satisfaite.
for while repeat
Commandes if et if/else
Les commandes if et if/else sont utiles pour:
- Vérifier s'il y a des problèmes ou le non-respect de conditions.
- Traiter des sous-ensembles de vos données de façons différentes.
- Vérifier l'existence d'un fichier ou d'une variable.
Commande if
if(condition) { expression }
Commande if else
if(condition) { expression 1 } else { expression 2 }
Comment peut-on tester plus qu'une condition?
ifetif elsetestent une seule condition- On peut aussi utiliser la commande
ifelsepour:- tester un vecteur de conditions;
- effectuer une opération seulement selon certaines conditions.
Par exemple,
a <- 1:10 ifelse(a > 5, "oui", "non")
a <- (-4):5 sqrt(ifelse(a >= 0, a, NA))
Commandes if else nichées
if (test_expression1) { statement1 } else if (test_expression2) { statement2 } else if (test_expression3) { statement3 } else { statement4 }
Exercice 1
Minou <- "chat" Pitou <- "chien" Filou <- "chat" animaux <- c(Minou, Pitou, Filou)
1. Utilisez une commande “if” pour afficher “meow” si Animal a la valeur “chat”.
2. Utilisez une commande if/else pour afficher “woof” si Animal a la valeur “chien” et “meow” sinon. Essayez en d'abord la valeur Animal=“chien” et ensuite “lion”.
3. Utilisez la commande ifelse pour afficher “woof” si les animaux sont des chiens et “meow” pour les chats.
Attention à la syntaxe!
Les accolades { } sont utilisées pour indiquer à R que l'expression continue sur plusieurs lignes et est à exécuter au complet. Par exemple, essayez:
if ((2 + 1) == 4) print("Les maths, c'est logique!.") else print("Houston, on a un problème.")
La commande else ne fonctionne pas, parce que R évalue la première ligne sans reconnaître que votre expression continue sur la deuxième ligne.
Utilisez plutôt:
if ((2 + 2) == 4) { print("Les maths, c'est logique!") # R n'évalue pas encore cette expression puisque l'accolade n'est pas fermée. } else { print("Houston, on a un problème.") } # Comme toutes les accolades sont fermées, R va évaluer les commandes en entier.
Rappel: opérateurs logiques
| == | égal à |
| != | pas égal à |
| !x | non x |
| < | plus petit que |
| < = | plus petit que ou égal à |
| > | plus grand que |
| >= | plus grand que ou égal à |
| x & y | x ET y |
| x|y | x OU y |
| isTRUE(x) | est-ce que X est vrai? |
Itération
Une boucle permet de répéter une ou des opérations.
Les boucles sont utiles pour:
- faire quelque chose pour chaque élément d'un objet.
- faire quelque chose jusqu'à la fin des données à traiter.
- faire quelque chose pour chaque fichier dans un répertoire.
- faire quelque chose qui peut échouer, jusqu'à ce que ça fonctionne.
- faire des calculs itératifs jusqu'à convergence.
Boucles "for"
La boucle for exécute un nombre fixe d'itérations d'un bloc de commande(s) .
for (variable in séquence) { expression }
La lettre “i” peut être remplacée par n'importe quelle nom de variable et la séquence peut être à peut prêt n'importe quoi, même une liste de vecteurs.
Essayez:
for (a in c("Bonjour", "programmeurs", "en R")) { print(a) } for (z in 1:30) { a <- rnorm(n = 1, mean = 5, sd = 2) # obtenir une valeur aléatoire provenant d'une distribution normale avec une moyenne de 5 et un écart type de 2. print(a) } elements <- list(1:3, 4:10) for (element in elements) { print(element) }
Dans l'exemple qui suit, R évaluerait l'expression 5 fois:
Par exemple:
for (i in 1:5) { print(i) }
Dans cet exemple, la variable m est remplacé successivement par chaque chiffre de 1 à 10, jusqu'au dernier élément de la séquence.
for(m in 1:10) { print(m*2) }
for(m in 1:5) { print(m*2) }
for(m in 6:10) { print(m*2) }
x <- c(2,5,3,9,6) count <- 0 for (val in x) { if(val %% 2 == 0) { count = count+1 } } print(count)
Les boucles for sont souvent utilisées pour exécuter des opérations successivement sur un jeu de données. Nous utiliserons ces boucles pour évaluer des fonctions sur le jeu de données CO2, qui est intégré dans R. Notez que c'est le même jeu de données utilisé pour l'atelier 2.
data(CO2) # ceci charge le jeu de données dans R for (i in 1:length(CO2[,1])) { # pour chaque ligne du jeu de données CO2 print(CO2$conc[i]) # affiche les concentrations de CO2 } for (i in 1:length(CO2[,1])) { # pour chaque ligne du jeu de données CO2 if(CO2$Type[i] == "Quebec") { # si le type est "Quebec" print(CO2$conc[i]) # affichez les concentrations de CO2 } } }
Truc 1. Pour exécuter une boucle sur chaque ligne d'un jeu de données, on utilise la fonction nrow().
for (i in 1:nrow(CO2)) { # pour chaque ligne du jeu de données CO2 print(CO2$conc[i]) # affichez les concentrations de CO2 }
Truc 2. On peut itérer des opérations sur une seule colonne.
for (i in CO2$conc) { # pour chacune des valeurs de concentration de CO2 print(i) # afficher cette valeur }
Truc 3. La partie “expression” de la boucle peut contenir plusieurs lignes de commandes différentes.
for (i in 4:5) { # pour i de 4 à 5 print(colnames(CO2)[i]) print(mean(CO2[,i])) # afficher les moyennes de cette colonne }
Boucles "for" nichées
Dans certains cas, vous voudrez peut-être utiliser des boucles nichées pour accomplir une tâche. Dans ce cas, il est important d'utiliser un nom de variable d'itération différent pour chaque boucle (ici on utilise i et n).
for (i in 1:5) { for (n in 1:5) { print (i*n) } }
Encore mieux: utiliser la famille "apply()"
La famille de fonctions apply() consiste de fonctions vectorisées qui réduisent le besoin de créer des boucles de façon explicite.
apply() est utilisé pour appliquer des fonctions sur une matrice.
(height <- matrix(c(1:10, 21:30), nrow = 5, ncol = 4)) } apply(X = height, MARGIN = 1, FUN = mean) ?apply
lapply()
lapply() applique une fonction sur chaque élément d'une liste.
lapply() peut aussi être utilisé avec d'autres objects, comme des trames de données (“dataframe”), listes, ou vecteurs.
La sortie est une liste (d'où le “l” dans lapply) ayant le même nombre d'éléments que l'objet d'entrée.
SimulatedData <- list( SimpleSequence = 1:4, Norm10 = rnorm(10), Norm20 = rnorm(20, 1), Norm100 = rnorm(100, 5)) # Applique mean() sur chaque élément de la liste lapply(SimulatedData, mean)
sapply()
sapply() est une fonction 'wrapper' pour lapply() qui produit une sortie simplifiée en vecteur, au lieu d'une liste.
SimulatedData <- list(SimpleSequence = 1:4, Norm10 = rnorm(10), Norm20 = rnorm(20, 1), Norm100 = rnorm(100, 5)) # Apply mean to each element of the list sapply(SimulatedData, mean)
mapply()
mapply() est une version multivariée de sapply().
mapply() applique une fonction premièrement sur le premier élément de chaque argument, et ensuite sur le deuxième élément, et ainsi de suite.
lilySeeds <- c(80, 65, 89, 23, 21) poppySeeds <- c(20, 35, 11, 77, 79) # Output mapply(sum, lilySeeds, poppySeeds)
tapply()
tapply() applique une fonction sur des sous-ensembles d'un vecteur.
tapply() est surtout utilisé quand un jeu de données contient différents groupes (ou niveaux/facteurs), et qu'on veut appliquer une fonction sur chaque groupe.
head(mtcars) # obtient la moyenne de hp par groupe de cylindres tapply(mtcars$hp, mtcars$cyl, FUN = mean)
Exercice 2
Vous avez réalisé que votre outil pour mesurer le l'absorption de CO2 n'était pas bien calibré aux sites situés au Québec, et toutes les mesures sont donc deux unités trop élevées.
- Utilisez une boucle pour corriger les mesures pour tous les sites aux Québec.
- Utilisez la famille de fonctions
apply()pour calculer la moyenne de l'absorption de CO2 dans les deux groupes de sites.
Assurez-vous de bien recharger le jeu de données pour ainsi travailler avec les données originales pour le reste de l'exercice:
data(CO2)
Modifications aux boucles
Normalement, les itérations s'exécutent successivement jusqu'à la dernière.
Il est parfois intéressant d'arrêter l'exécution de la boucle quand une certaine condition est satisfaite ou quand l'itération a atteint un élément.
On peut aussi arrêter l'exécution de l'itération courante pour passer à la boucle suivante.
Pour ceci, on introduit break, while, et next.
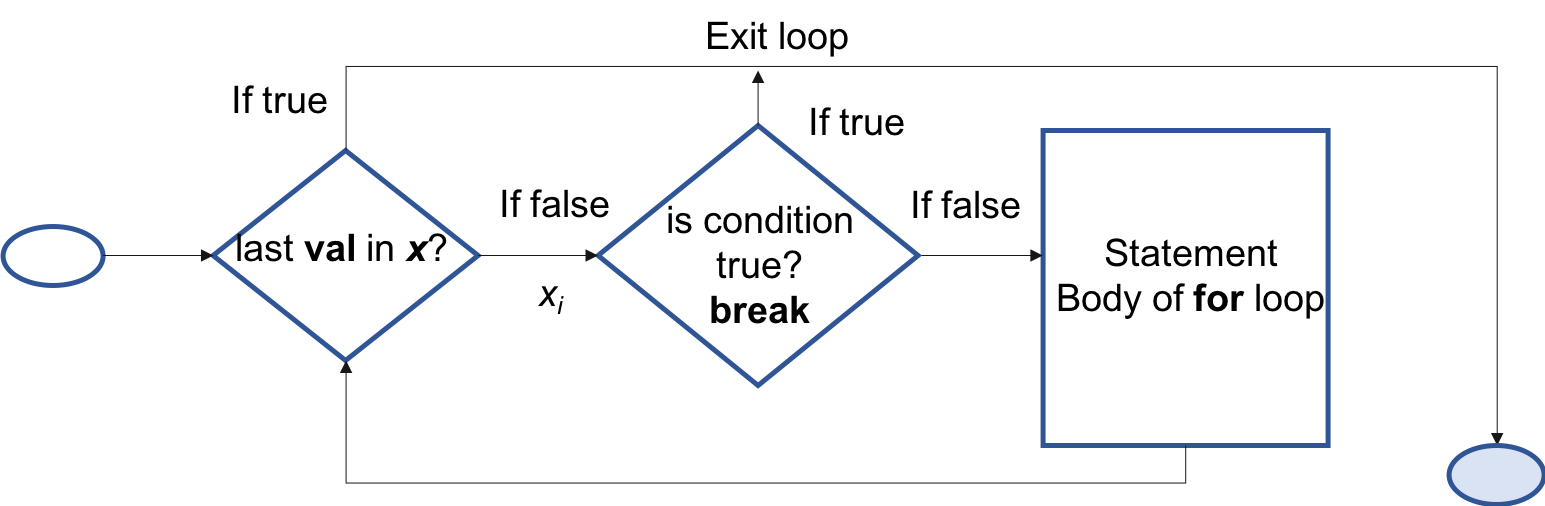
Modifications aux boucles: "break"
for(val in x) { if(condition) { break } statement }
Modifications aux boucles: "next"
for(val in x) { if(condition) { next } statement }

Par exemple, on veut afficher les concentrations de CO2 pour les traitements “chilled” et garder le compte du nombre total d'itérations accomplies.
count <- 0 # la valeur de count est mise à zéro pour pouvoir modifier la valeur dans la boucle. for (i in 1:length(CO2[,1])) { if (CO2$Treatment[i] == "nonchilled") next # Passer à l'itération suivante si c'est "nonchilled" count <- count + 1 print(CO2$conc[i]) } print(count) # Les fonctions count et print ont été exécutées 42 fois. sum(CO2$Treatment == "nonchilled")
Modifications aux boucles: "break"
Ceci pourrait être écrit de façon équivalente en utilisant une boucle repeat et break:
count <- 0 i <- 0 repeat { i <- i + 1 if (CO2$Treatment[i] == "nonchilled") next # sauter cette itération count <- count + 1 print(CO2$conc[i]) if (i == length(CO2[,1])) break # arrêter l'itération } print(count)
Modifications aux boucles: "while"
On pourrait écrire ceci avec une boucle while.
i <- 0 count <- 0 while (i < length(CO2[,1])) { i <- i + 1 if (CO2$Treatment[i] == "nonchilled") next # sauter cette itération count <- count + 1 print(CO2$conc[i]) } print(count)
Exercice 3
Vous venez de réaliser que votre outil pour mesurer la concentration ne fonctionne pas correctement.
Aux sites situés au Mississippi, les concentrations de moins de 300 sont bien mesurés, mais les concentrations de plus de 300 étaient surestimées par 20 unités.
Votre mission est d'écrire une boucle pour corriger ces mesures pour les sites du Mississippi.
Truc: Assurez-vous que vous travaillez avec les données originales pour le reste de l'exercice:
data(CO2)
Visualization de données avec "for" et "if"
Nous voulons créer un graphique à partir des données de concentration et absorption où chaque point est associé à un type (Québec ou Mississippi) et un traitement (“chilled” et “nonchilled”), et nous voulons représenter ces points différemment.
head(CO2) # Voir les données unique(CO2$Type) unique(CO2$Treatment) # Créer le graphique dans lequel chaque type et traitement a une couleur différente plot(x=CO2$conc, y=CO2$uptake, type="n", cex.lab=1.4, xlab="CO2 concentration", ylab="CO2 uptake") # Type "n" dit à R de ne pas créer le graphique for (i in 1:length(CO2[,1])) { if (CO2$Type[i] == "Quebec" & CO2$Treatment[i] == "nonchilled") { points(CO2$conc[i], CO2$uptake[i], col="red",type="p") } if (CO2$Type[i] == "Quebec" & CO2$Treatment[i] == "chilled") { points(CO2$conc[i], CO2$uptake[i], col="blue") } if (CO2$Type[i] == "Mississippi" & CO2$Treatment[i] == "nonchilled") { points(CO2$conc[i], CO2$uptake[i], col="orange") } if (CO2$Type[i] == "Mississippi" & CO2$Treatment[i] == "chilled") { points(CO2$conc[i], CO2$uptake[i], col="green") } }
Exercice 4
Créez un graphique montrant les concentrations en fonction de l'absorption et où chaque plante est représentée par des points de couleurs différentes.
Bonus: Essayez de le faire avec une boucle nichée!
Écrire des fonctions
Pourquoi créer ses fonctions?
Le gros du travail dans R est fait par des fonctions. Elles sont utiles pour:
- répéter une même tâche mais en changeant ses paramètres
- rendre votre code plus lisible
- rendre votre code plus facile à modifier et à maintenir
- partager du code entre différentes analyses
- partager votre code avec d'autres personnes
- modifier les fonctionalités par défaut de R
Mais qu'est ce qu'une fonction au juste? Une fonction, c'est essentiellement une boîte noire qui transforme des données. Elle prend en entrée des valeurs - appelées arguments -, utilise du code R pour les traiter et renvoie optionnellement une valeur de retour.
Syntaxe d'une fonction
nom_de_la_fonction <- function(argument1, argument2, ...) { expression... # Ce que la fonction fait return(value) # Optionnel, pour accéder au résultat de la fonction }
Arguments d'une fonction
Les arguments sont les données fournies en entrée à votre fonction. Il s'agit de l'information dont votre fonction a besoin pour opérer correctement.
Une fonction peut avoir entre 0 et une infinité d'arguments.
Par exemple, créons une fonction qui prend un premier nombre (number1), l'additionne à un second (number2), multiplie le résultat par un troisième nombre (number3) et enfin affiche le résultat.
operations <- function(number1, number2, number3) { result <- (number1 + number2) * number3 print(result) } operations(1, 2, 3) operations(17, 23, 2)
Exercice 5
En utilisant ce que vous avez vu précédemment sur les structures de contrôle, créez une fonction appelée print_animal qui prend un animal en argument et donne les résultats suivants:
Scruffy <- "dog" Paws <- "cat" print_animal(Scruffy) [1] "woof" print_animal(Paws) [1] "meow"
Valeurs par défaut dans une fonction
Les arguments peuvent également être optionnels, auquel cas on peut leur donner une valeur par défaut.
Ceci peut s'avérer utile si l'on prévoit d'utiliser fréquemment une fonction avec les mêmes paramètres pour éviter d'avoir à les réécrire à chaque fois, mais si l'on veut tout de même garder la possibilité de changer leur valeur si nécessaire.
operations <- function(number1, number2, number3 = 3) { result <- (number1 + number2) * number3 print(result) } operations(1, 2, 3) # est équivalent à operations(1, 2) operations(1, 2, 2) # on peut toujours changer la valeur de number3
Argument "..."
L'argument spécial “…” vous permet de passer des arguments à une autre fonction utilisée à l'intérieur de votre fonction.
Par exemple, créons une fonction à partir de notre exemple précédent où nous traçons l'absorption de CO2 en fonction de la concentration. Nous allons tracer nos graphes avec deux couleurs différentes selon la région. Ici, les paramètres de plot() et points() seront passés via “…”.
plot.CO2 <- function(CO2, ...) { plot(x=CO2$conc, y=CO2$uptake, type="n", ...) # On utilise ... pour passer les arguments a plot(). for (i in 1:length(CO2[,1])){ if (CO2$Type[i] == "Quebec") { points(CO2$conc[i], CO2$uptake[i], col="red", type="p", ...) # idem pour points() } else if (CO2$Type[i] == "Mississippi") { points(CO2$conc[i], CO2$uptake[i], col="blue", type="p", ...) # idem pour points() } } } plot.CO2(CO2, cex.lab=1.4, xlab="CO2 concentration", ylab="CO2 uptake") plot.CO2(CO2, cex.lab=1.4, xlab="CO2 concentration", ylab="CO2 uptake", pch=20)
L'argument spécial … autorise l'utilisateur à entrer un nombre indéfini d'arguments. La valeur de chaque argument devra alors être récupérée manuellement. Par exemple, créons une fonction somme qui accepte un nombre indéfini d'arguments.
sum2 <- function(...){ args <- list(...) result <- 0 for (i in args) { result <- result + i } return (result) } sum2(2, 3) sum2(2, 4, 5, 7688, 1)
Valeurs de retour
La dernière expression évaluée dans une fonction sera la valeur de retour, même sans la fonction return().
myfun <- function(x) { if (x < 10) { 0 } else { 10 } } myfun(5) myfun(15)
Utiliser return() peut être utile si la boucle doit terminer tôt, sortir de la fonction, et sortir une valeur.
simplefun1 <- function(x) { if (x<0) return(x) }
Une seule valeur de retour peut être renvoyée par une fonction. Si vous désirez renvoyer plus d'un objet, vous devez utiliser des objets tels que des listes ou des dataframes.
Par ailleurs, il est important de noter que l'execution de la fonction se termine dès qu'elle atteint le mot clé return().
simplefun2 <- function(x, y) { z <- x + y return(list("result" = z, "x" = x, "y" = y)) } simplefun2(1, 2)
Exercice 6
En utilisant ce que vous avez appris jusqu'ici sur les fonctions et les structures de contrôle, créez une fonction bigsum qui prend deux arguments a et b et :
- sort 0 si la somme de a et b est strictement inférieure à 50
- sinon, sort la somme de a et b
Accessibilité des variables
Il est essentiel de pouvoir situer nos variables, et de savoir si elles sont définies et accessibles.
- Les variables définies à l'intérieur d'une fonction ne sont pas accessibles en dehors de la fonction!
- Les variables définies à l'extérieur d'une fonction sont accessibles à l'intérieur. Cependant, ce n'est JAMAIS une bonne idée de les utiliser à l'intérieur, car votre fonction pourrait arrêter de fonctionner si la variable est effacée.
var1 <- 3 # var1 est définie à l'extérieur de la fonction vartest <- function() { a <- 4 # a est définie a l'intérieur print(a) # affiche a print(var1) # affiche var1 } a # affiche a. Ceci ne fonctionne pas, # a est seulement visible dans la fonction vartest() # vartest() affiche a et var1 rm(var1) # supprime var1 vartest() # la fonction ne fonctionne plus, # car var1 n'existe plus
Utilisez donc des arguments!!
Dans une fonction, les noms d'arguments remplaceront les noms des autres variables.
var1 <- 3 # var1 est définie à l'extérieur de la fonction vartest <- function(var1) { print(var1) # affiche var1 } vartest(8) # Dans notre fonction, var1 est maintenant notre argument et prend sa valeur var1 # var1 a toujours la meme valeur
Faites très attention lorsque vous créez des variables à l'intérieur d'une condition, car la variable pourrait ne jamais être créée et causer des erreurs parfois imperceptibles.
a <- 3 if (a > 5) { b <- 2 } a + b # Erreur! b n'existe pas!
Si b avait déjà une valeur différente assignée dans l'environnement, on aurait un gros problème!
R ne trouverait pas d'erreur, et la valeur de a + b serait entièrement différente!
Bonnes pratiques
Voici quelques conseils de programmation qui peuvent:
- Vous faciliter la vie
- Vous aider à avoir un code plus lisible
- Faciliter le partage et la réutilisation de votre code
- Réduire le temps que vous passeriez à essayer de comprendre votre code.
Gardez un code beau et propre
Voici quelques trucs pour vous aider:
- Mettez des espaces avant et après vos opérateurs
- Utilisez toujours le même opérateur d'assignation.
←est préférable (=fonctionne, mais ne changez pas entre les deux) - Utilisez des crochets pour encadrer vos structures de contrôle, même si c'est seulement pour une ligne.
- À l'intérieur des crochets, faites un alinéa d'au moins deux espaces pour chaque ligne de code.
- Les crochets de fermeture occupent généralement leur propre ligne, sauf s'ils précèdent une condition else.
- Définissez chaque variable sur sa propre ligne
Voici un exemple de code qui est est mal espacé et aligné, et donc difficile à lire:
a<-4;b=3 if(a<b){ if(a==0)print("a zero") } else { if(b==0){print("b zero")} else print(b)}
Voici une version plus facile à lire:
a <- 4 b <- 3 if(a < b){ if(a == 0) { print("a zero") } } else { if(b == 0){ print("b zero") } else { print(b) } }
Utilisez des fonctions pour simplifier le code
Écrivez vos propres fonctions:
- Quand une portion du code est répété à plus de deux reprises dans ton script.
- Quand seulement une partie du code change et inclut des options pour différents arguments.
Ceci vous aidera à réduire le nombre d'erreurs de copier/coller, et réduira le temps passé à les corriger et facilitera les modifications futures.
Par exemple, modifions l'exemple de l'exercice 3 et supposons que toutes les absorptions de CO2 du Mississipi étaient surestimées de 20 et que celles du Québec étaient sous-estimées de 50. Nous pourrions écrire ceci:
for (i in 1:length(CO2[,1])) { if(CO2$Type[i] == "Mississippi") { CO2$conc[i] <- CO2$conc[i] - 20 } } for (i in 1:length(CO2[,1])) { if(CO2$Type[i] == "Quebec") { CO2$conc[i] <- CO2$conc[i] + 50 } }
Ou alors:
recalibrate <- function(CO2, type, bias) { for (i in 1:nrow(CO2)) { if(CO2$Type[i] == type) { CO2$conc[i] <- CO2$conc[i] + bias } } return(CO2) } newCO2 <- recalibrate(CO2, "Mississipi", -20) newCO2 <- recalibrate(newCO2, "Quebec", +50)
Noms de fonctions informatifs
Ceci aide à voir au premier coup d'oeil qui fait quoi. De plus, choisir des noms courts évite les fautes de frappe.
Voici ce à quoi notre exemple précédent pourrait ressembler avec un nom vague.
rc <- function(c, t, b) { for (i in 1:nrow(c)) { if(c$Type[i] == t) { c$uptake[i] <- c$uptake[i] + b } } return(c) }
Utilisez des commentaires
Ajoutez des commentaires pour décrire tout ce que votre code fait, que ce soit le but de la fonction, comment utiliser ses arguments, ou une description détaillée de la fonction étape par étape.
## Recalibre le jeu de données CO2 en modifiant l'absorption de CO2 ## d'une valeur fixe selon la region # Arguments # CO2: le jeu de donnees CO2 # type: le type de données à recalibrer ("Mississippi" ou "Quebec") # bias: la quantité à ajouter ou soustraire de l'absorption. recalibrate <- function(CO2, type, bias) { for (i in 1:nrow(CO2)) { if(CO2$Type[i] == type) { CO2$uptake[i] <- CO2$uptake[i] + bias } } return (CO2) }