AVIS IMPORTANT
Depuis l'automne 2021, ce wiki a été discontinué et n'est plus activement développé.
Tout le matériel mis à jour et les annonces pour la série d'ateliers R du CSBQ se trouvent maintenant sur le site web de la série d'ateliers R du CSBQ. Veuillez mettre à jour vos signets en conséquence afin d'éviter les documents périmés et/ou les liens brisés.
Merci de votre compréhension,
Vos coordonnateurs de la série d’ateliers R du CSBQ.
Ateliers R du CSBQ
Cette série de 10 ateliers guide les participants à travers les étapes requises afin de maîtriser le logiciel R pour une grande variété d’analyses statistiques pertinentes en recherche en biologie et en écologie. Ces ateliers en libre accès ont été créés par des membres du CSBQ à la fois pour les membres du CSBQ et pour la grande communauté d’utilisateurs de R.
Le contenu de cet atelier a été révisé par plusieurs membres du CSBQ. Si vous souhaitez y apporter des modifications, veuillez SVP contacter les coordonnateurs actuels de la série, listés sur la page d'accueil
Atelier 2 : Charger et manipuler des données
Développé par : Johanna Bradie, Vincent Fugère, Thomas Lamy
Résumé : Durant cet atelier, vous apprendrez comment charger et visualiser des données dans R. Vous apprendrez certaines fonctions permettant d’examiner vos données et vous apprendrez comment corriger certaines erreurs communes se produisant lors du chargement des données. Finalement, vous verrez l’importance de sauvegarder votre travail dans un script R, soit un fichier texte contenant tous vos codes et permettant de refaire toutes vos analyses en appuyant sur une seule touche (ou deux, ou trois peut-être…). Ensuite, nous présenterons tidyr et dplyr pour vous aider à transformer et à gérer le format de vos données ainsi que pour appliquer des fonctions simples ou complexes sur des sous-groupes de vos jeux de données. Cet atelier s’avérera utile non seulement pour les participants suivant notre série d’ateliers, mais également pour ceux et celles d’entre vous connaissant déjà le langage R et cherchant à maîtriser des outils plus avancés pour l'analyse et la visualisation de vos données.
Lien vers la nouvelle présentation Rmarkdown
S'il vous plaît essayez-la et dites aux coordonnateurs des ateliers R ce que vous en pensez!
Lien vers l'ancienne présentation Prezi
Téléchargez les scripts et les données requis pour cet atelier :
Objectifs d'apprentissage
- Créer un projet RStudio
- Écrire un script dans R
- Charger, explorer et enregister des données
- Manipuler des jeux de données avec tidyr, dplyr, maggritr
Les projets RStudio
Qu-est-ce qu'un projet RStudio?
- Les projets RStudio permettent l'organisation de son travail et l'accès facile à tous les fichiers requis pour une analyse.
- Tout fichier, script, et documentation utilisé pour une analyse sont reliés et regroupés ensemble dans un même projet.
L'utilisation de projets RStudio facilite aussi la reproducibilité et le partage de données, de scripts, et de leur documentation.
Créer un projet dans RStudio
Pour créer un projet dans RStudio, allez dans le menu Fichier puis sélectionnez “Nouveau projet”.
Organiser ses fichiers
Un projet = un dossier!
Préparer des données pour l'importation dans R
- Vous devriez enregistrez les fichiers à importer dans R en tant que fichiers “comma separated values” (.csv)
- Les fichiers .csv peuvent être créés par presque toutes les applications (Excel, GoogleDocs, LibreOffice, etc.)
- Fichier → Enregistrer sous: .csv
Bien nommer les fichiers
- Oui:
- rawDatasetAgo2017.csv
- co2_concentrations_QB.csv
- 01_figIntro.R
- Non:
- final.csv (Pas informatif!)
- safnnejs.csv (C'est n'importe quoi!)
- 1-4.csv (Éviter d'utiliser des chiffres!)
- Dont.separate.names.with.dots.csv (Peut causer des erreurs de lecture de fichier!)
Bien nommer les variables
- Utilisez des noms de variables courts et informatifs (i.e. “Temps_1” au lieu de “Temps de la premiere mesure”)
- Oui: “Mesure”, “NomEspece”, “Site”
- Les valeurs des colonnes doivent correspondre à l'usage prévu
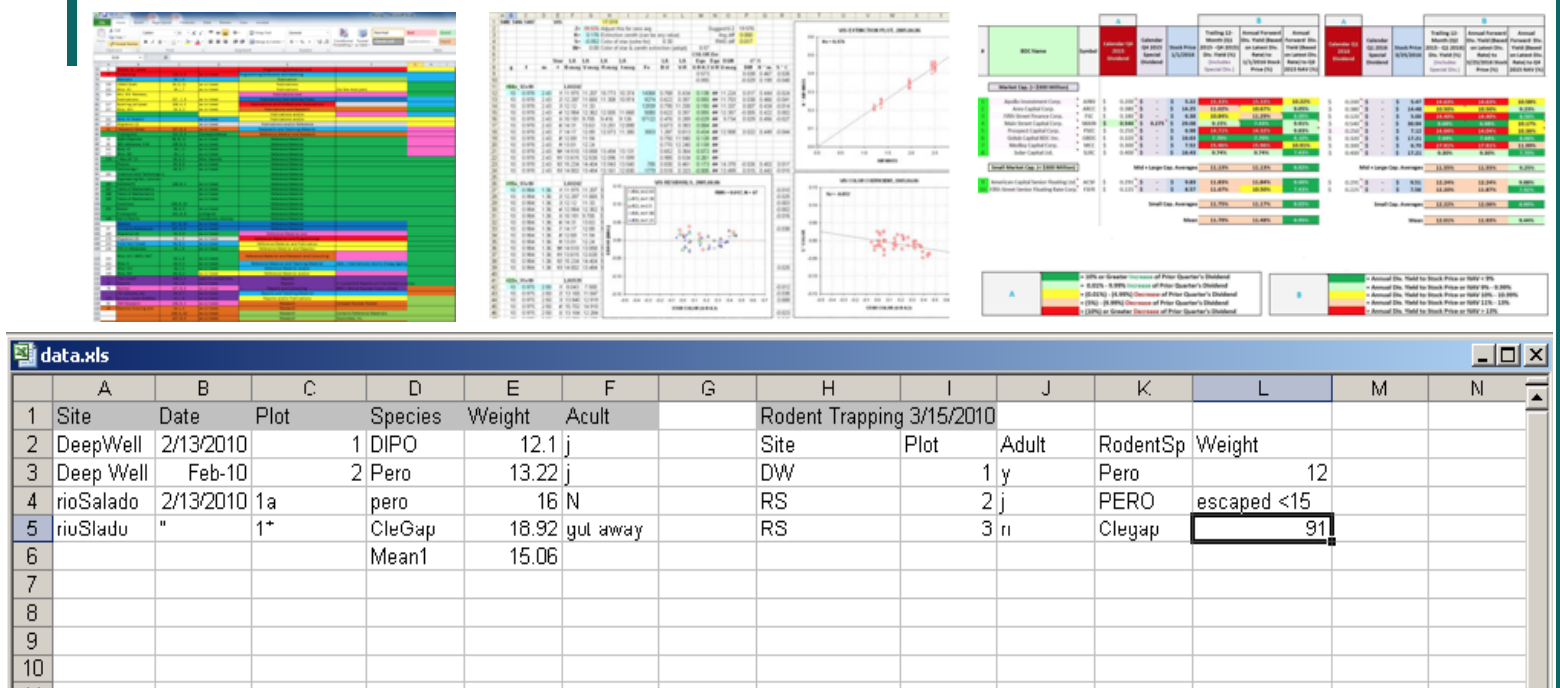
Conseils pour la préparation de jeux de données
- Pas de texte dans les colonnes de mode numérique (ceci inclut les espaces !)
- Identifiez les valeurs manquantes par NA (“not available”) - notez le majuscule!
- Faites attention aux erreurs typographiques!
- Évitez les valeurs numériques pour les variables n'ayant pas vraiment de valeur numérique (i.e. individu, réplicat, traitement)
- Par exemple, identifiez vos individus par “A,B,C” ou “ind1,ind2,ind3” au lieu de “1,2,3”
- Utilisez un format uniforme pour les dates, les chiffres, etc.
- N'ajoutez pas de notes, d'en-têtes supplémentaires, ou de cellules fusionnées!
- Seulement une variable par colonne!
Exemples de mauvaises habitudes
Il est possible de faire toute la préparation des données dans R. Les avantages sont :
- On économise du temps pour les grosses bases de données
- On préserve les données d'origine
- On peut basculer entre les modes “long” et “large” très facilement (plus de détails plus tard)
- Pour des informations complémentaires, consultez la page suivante : https://www.zoology.ubc.ca/~schluter/R/data/
Écrire un script dans R
Un script R est un fichier texte contenant toutes les commandes nécessaires pour réaliser un projet. Une fois écrit et enregistré, votre script R vous permettra d'apporter des changements et de refaire des analyses avec un minimum d'effort.
Afin d'utiliser un script dans RStudio, il suffi simplement de surligner une commande et appuyer sur “Run” ou appuyer sur “command-enter” (Mac) ou “ctrl-enter” (PC).
Commandes & Commentaires
Utilisez le symbole '#' pour insérer des commentaires au sein d'un script. Ceci indique à R d'ignorer tout ce qui se trouve à la suite du symbole '#' lors de l'exécution de commandes.
Puisque les commentaires sont ignorés par R lors de l'exécution de scripts, il est possible de se laisser des notes ou d'indiquer à des collaborateurs ce qui a été fait. Un script enrichi de commentaires est une bonne façon de collaborer à une science reproductible et permet de comprendre comment R fonctionne.
# Ceci est un commentaire, pas une commande !
En-têtes
Il est recommandé d'ajouter des commentaires au début de chaque script sous forme d'en-tête en indiquant quelques informations essentielles : nom du projet, auteur, date et version de R.
- Exemple d'en-tête
## Atelier R du CSBQ ## ## Atelier 2 - Charger et manipuler des données ## Auteur: Centre de la science de la biodiversité du Québec ## Date: Automne 2014 ## R version 2.15.0
En-têtes de section
Vous pouvez utiliser quatre symboles '#' de suite pour créer un en-tête de section. C'est utile pour organiser un script.
Par exemple :
#### Chargement du fichier de données ####
RStudio affiche une petite flèche juste à côté du numéro de ligne où l'en-tête de section a été créé. Si vous cliquez sur la flèche, cette partie du script sera cachée.
Vous pouvez aussi naviguer entre sections en utilisant le menu trouvé en bas de la fenêtre du script.
Nettoyage
C'est une bonne habitude de maintenir son espace de travail propre en effaçant la mémoire de R ! À l'aide d'une simple commande, il est possible d'éviter plusieurs erreurs telles qu'utiliser un vieux jeu de données qui serait resté dans l'espace de travail. La commande rm(list=ls()) efface la mémoire.
On peut tester cette commande en ajoutant des objets dans l'espace de travail pour mieux comprendre comment rm(list=ls()) fonctionne.
- |
A<-"Test" # On crée un objet "A". A <- "Test" # C'est préférable de mettre des espaces avant et après "<-". A = "Test" # = peut aussi être utilisé comme opérateur d'assignation #Tel que vu au premier atelier, il est préférable d'utiliser "<-" comme opérateur d'assignation plutôt que "=". A rm(list=ls()) A
Petit rappel important
- R est prêt à exécuter une commande lorsque vous voyez le chevron
>affiché dans la console. Si le chevron n'apparaît pas, c'est que la commande est incomplète : R attend que vous complétiez la commande. Appuyez sur 'ESC' pour sortir de cette commande. - R est sensible à la casse, i.e. “A” est différent de “a”.
Charger, explorer et enregistrer des données
Répertoire de travail
Vous devez indiquer à R le répertoire où se trouvent les fichiers de données afin de les charger. Vous pouvez voir quel répertoire R utilise avec la fonction getwd()“
Lorsque vous chargez un script, R définit le répertoire de travail comme étant le dossier qui contient le script.
Afin de spécifier le chemin d'accès du répertoire avec la fonction setwd(), utilisez le ”/“ pour séparer les dossiers, sous-dossiers et noms de fichiers. Vous pouvez aussi cliquer sur session / Définir le répertoire / Choisir le répertoire.
Afficher le contenu du répertoire de travail
La fonction dir() affiche le contenu du répertoire de travail.
Vous pouvez vérifier:
- Si le fichier que vous voulez ouvrir se trouve dans le répertoire de travail ou non
- L'orthographe du nom du fichier (e.g. 'monfichier.csv' au lieu de 'MonFichier.csv')
Importer un jeu de données
Utilisez la fonction read.csv() pour importer des données provenant d'un fichier .csv dans R.
- |
CO2<-read.csv("CO2_good.csv") # Cette commande crée un objet nommé CO2 en chargeant les données # du fichier nommé "CO2_good.csv".
Cette commande spécifie que vous créez un objet nommé “CO2” en lisant un fichier .csv nommé “CO2_good.csv”. Ce fichier doit se trouver dans votre répertoire de travail.
Il est aussi possible d'utiliser la fonction file.choose() pour charger un fichier de manière interactive.
- |
CO2<-read.csv(file.choose())
Rappelez-vous que le point d'interrogation peut être utilisé pour trouver de l'aide sur une fonction.
La page d'aide permet de voir qu'il y a un argument header qui permet de spécifier si la première ligne du fichier contient le nom des colonnes. Inscrire header=TRUE indique à R que la première ligne du tableau contient les noms des colonnes.
NOTE : Si vous utilisez un système d'exploitation en français ou un éditeur CSV, il est possible que vous deviez utiliser la fonction read.csv2() au lieu de la fonction read.csv() pour importer correctement un fichier.
Prenez note que RStudio montre maintenant le jeu de donné CO2 dans ton espace de travail. L'espace de travail inclut tous les objets créés pendant ta session R.
Explorer le jeu de données
Le jeu de données “CO2” contient des mesures répétées d'absorption de CO2 prises sur six plantes du Québec et six plantes du Mississippi à différentes concentrations de CO2 ambiant. La moitié des plantes de chaque région a subi un traitement de refroidissement la veille du début de l'expérience.
Voici quelques fonctions utiles pour visualiser le jeu de données importé :
| CO2 | Affiche le tableau de données complet dans la console |
| head(CO2) | Affiche les premières lignes du tableau de données |
| names(CO2) | Affiche le nom des colonnes du tableau de données |
| attributes(CO2) | Affiche les attributs du tableau de données |
| ncol(CO2) | Affiche le nombre de colonnes du tableau de données |
| nrow(CO2) | Affiche le nombre de lignes du tableau de données |
| summary(CO2) | Calcule quelques statistiques de base sur les variables |
| str(CO2) | Affiche la structure du tableau de données |
La fonction str() est très utile pour identifier le type/mode de chaque colonne (e.g. afin de vérifier que les facteurs sont enregistrés comme facteurs et que les données numériques sont de type numérique ou nombre entier). Plusieurs problèmes sont fréquents lors de l'importation des données :
- Les facteurs apparaissent comme des chaînes de caractères (et vice versa)
- Les facteurs ont trop de niveaux à cause d'une erreur de frappe
- Les données numériques sont stockées sous forme de chaînes de caractères à cause d'une erreur de frappe (e.g. un espace de trop ou utiliser la virgule au lieu du point pour indiquer les décimales)
Exercice
Chargez les données de nouveau en utilisant le script suivant :
Vérifiez la structure des données avec la fonction str(). Quel est le problème ? Chargez les données de nouveau, mais cette fois-ci avec l'argument header=TRUE avant de continuer.
Rappel de l'atelier 1 : Accéder aux données
Les données peuvent être extraites d'un jeu de données de plusieurs façons. Considerons un jeu de données nommé mydata. Les crochets permettent d'extraire le contenu d'une cellule.
- |
mydata[2,3] # extrait le contenu de la deuxième ligne / troisième colonne
Si le numéro de la colonne est omis, toute la ligne est extraite.
- |
mydata[1,] # extrait le contenu de la première ligne
Si le numéro de la ligne est omis, toute la colonne est extraite. De même, le symbôle $ suivi d'un nom de colonne peut être utilisé.
- |
mydata[,1] # extrait le contenu de la première colonne mydata$Variable1 # extrait le contenu de la colonne "Variable1"
Changer les noms de variables
On peut changer le nom de variables (colonnes) dans R.
- |
# Créer une copie du jeu de données qu'on pourra modifier CO2copy <- CO2 # names() donne les noms des variables présents dans le jeu de données names(CO2copy) # Changer des noms anglais à des noms français names(CO2copy) <- c("Plante","Categorie", "Traitement", "conc","absortion")
Créer des nouvelles variables
On peut facilement créer et produire des nouvelles variables. Par exemple, la fonction paste() permet la concaténation de chaînes de caractères et de variables.
- |
# Créer un ID unique pour les échantillons avec la fonction ''paste()'' # Consultez ?paste et ?paste0 # N'oubliez pas d'utiliser "" pour les chaînes de caractères CO2copy$uniqueID <- paste0(CO2copy$Plante,"_", CO2copy$Categorie, "_", CO2copy$Traitement) # Observer les résultats head(CO2copy$uniqueID)
On peut aussi créer des nouvelles variables à partir de chiffres et d'opérations mathématiques!
- |
# Standardizer la variable "absortion" en valeurs relatives CO2copy$absortionRel = CO2copy$absortion/max(CO2copy$absortion) # Observer les résultats head(CO2copy$absortionRel)
Extraire un sous-ensemble d'un jeu de données
Il y a plusieurs façons d'extraire un sous-ensemble d'un jeu de données.
- |
# Continuons avec notre trame de données "CO2copy" ## Extraire un sous-ensemble par un nom de variable CO2copy[,c("Plante", "absortionRel")] # Extrait seulement les colonnes nommées "Plante" et "absortionRel". (N'oubliez pas ","!) ## Extraire un sous-ensemble de rangées CO2copy[1:50,] # Extraite les rangées de 1 à 50 ### Extraire un sous-ensemble correspondant à un facteur CO2copy[CO2copy$Traitement == "nonchilled",] # Extraire les observations correspondant seulement au traitement "nonchilled". ### Extraire un sous-ensemble correspondant à une condition numérique CO2copy[CO2copy$absortion >= 20, ] # Extraire les observations ayant une absortion plus grande ou égale à 20 ### Les conditions peuvent être complémentaires en utilisant "&" CO2copy[CO2copy$Traitement == "nonchilled" & CO2copy$absortion >= 20, ] # Nous avons fini de modifier la copie du jeu de données CO2copy. On peut alors l'effacer. CO2copy <- NULL
Consultez here pour voir les opérateurs logiques pouvant être utilisés pour extraire des sous-ensembles de données dans R.
Explorer les données
Pour faire une première exploration de données, on peut rapidement calculer des statistiques de base avec la fonction summary().
Plusieurs fonctions intégrées à R vous permettent d'obtenir de l'information supplémentaire sur vos données. Les fonctions mean(), sd(), hist(), et print() sont couramment utilisées.
- |
# Calculer la moyenne et l'écart type des données dans la colonne "conc" de l'objet CO2 meanConc<-mean(CO2$conc) # Calcule la moyenne de la colonne "conc" de l'objet "CO2" sdConc<-sd(CO2$conc) # Calcule l'écart-type de la colonne "conc"
# print() sort une valeure dans la console R print(paste(“la concentration moyenne est:”, meanConc)) print(paste(“l'écart type de la concentration est:”, sdConc))
# Produisez un histogramme to explore the distribution of “uptake” hist(CO2$uptake)
# Augmenter le nombre de classes pour mieux observer la distribution hist(CO2$uptake, breaks = 40) </code>
La fonction apply() est utilisée pour appliquer une fonction à plusieurs colonnes en même temps. Utilisez ?apply pour en apprendre plus.
Pour utiliser la fonction apply(), trois arguments doivent être spécifiés. Le premier argument est le jeu de données sur lequel on veut appliquer une fonction. Le deuxième argument spécifie si la fonction est appliquée aux lignes (MARGIN = 1) ou aux colonnes (MARGIN = 2). Le dernier argument spécifie la fonction à appliquer. Par exemple :
- |
apply(CO2[,4:5], MARGIN = 2, FUN = mean) # Calcule la moyenne des colonnes 4 et 5 du jeu de données CO2
Enregistrer son espace de travail
En enregistrant votre espace de travail, vous pouvez enregistrer les scripts et les objets qui sont actuellement chargés dans R. Si vous enregistrez votre espace de travail, vous pouvez chargez tous les objets même si vous avez utilisé la commande rm(list=ls()) afin de nettoyer tout ce qui se trouve dans l'espace de travail.
Utilisez la fonction save.image() pour enregistrer l'espace de travail :
- |
save.image(file="CO2_project_Data.RData") # L'espace de travail est enregistré rm(list=ls()) # Nettoyer l'espace de travail load("CO2_project_Data.RData") # Charger tout ce qui se trouvait dans l'espace de travail head(CO2) # Ça fonctionne! :)
Exporter le jeu de données
Si vous voulez enregistrer des données que vous avez créées ou modifiées dans R, utilisez la fonction write.csv(). Le fichier ainsi créé sera enregistré dans le répertoire de travail.
DÉFI: Utilisez vos propres données
Essayez de charger, explorer, et enregistrer vos propres données dans R. Est-ce que le chargement fonctionne ? Si ce n'est pas le cas, essayez d'arranger vos données dans Excel. Enregistrez vos données “arrangées” et essayez de les ouvrir dans R.
Réparer un jeu de données
Les jeux de données peuvent être désordonnés et incompatibles entre certains systèmes (Mac, Windows) ou entre ordinateurs.
Pratiquons-nous à réparer certains problèmes communs.
DÉFI: Réparer un jeu de données "endommagé"
Importez un jeu de données “endommagé” dans R et trouvez les problèmes.
- |
CO2<-read.csv("CO2_broken.csv") # On remplace l'ancien objet CO2 avec le contenu d'un nouveau jeu de données head(CO2) # C'est le désordre! CO2
- Vos données ressemblent probablement à ça !
- Réparez le jeu de données dans R
- Faites un essai avant de regarder la solution
- Entraidez-vous entre voisins et amusez-vous :)
Voici quelques fonctions qui peuvent vous aider :
read.csv()head()str()class()unique()levels()which()droplevels()
Note : Vous devez mettre le nom de l'objet entre parenthèses pour utiliser ces fonctions (i.e. head(CO2)).
Rappelez-vous que vous pouvez obtenir de l'aide en utilisant ”?“ (e.g. ?str).
Indice : Il y a quatre problèmes avec ce jeu de données!
Réponse #1
Réponse #2
Réponse #3
Réponse #4
Manipuler ses données avec tidyr, dyplr, maggritr
Utiliser "tidyr" pour réorganiser ses données
Pourquoi réorganiser ses données?
La réorganisation permet de modifier la structure des données tout en préservant l'information contenue dans le jeu de données. Plusieurs fonctions dans R exigent ou fonctionnent mieux avec une structure de données qui n'est pas nécessairement propice à la lecture par l'oeil humain.
En comparaison à l'agrégation, où plusieurs cellules sont regroupées pour créer un nouveau jeu de données (e.g. tableau des moyennes de plusieurs variables), la réorganisation maintient le même nombre de cellules. Le paquet tidyr permet de réorganiser nos jeu de données dans un format idéal pour nos tâches en aval, et ce à l'aide d'une syntaxe simple et logique.
Commençons par installer et charger le paquet tidyr :
- |
if(!require(tidyr)){install.packages("tidyr")} library(tidyr)
Données en longueur vs. en largeur
Un jeu de données en largeur contient une colonne pour chaque variable ou facteur inclus dans les données. Une rangée peut alors comprendre plusieurs observations différentes.
Un jeu de données en longueur contient une colonne par variable, où chaque rangée s'agit d'une observation unique. Le format “long” est plus “propre” (tidy en anglais), parce que les données sont plus facilement interprétées par R pour nos visualisations et nos analyses.
Le format de vos données dépend de vos besoins d'analyse et de visualisation, mais certains paquets et fonctions, incluant dplyr, lm(), glm(), et gam(), nécessitent des données en longueur. Le paquet ggplot2 peut utiliser des données en largeur pour des visualisations simples, mais fonctionne mieux avec des données en longueur pour des visualisations plus complexes (exemple à venir).
De plus, les données en format long peuvent être agrégées et réorganisées en format large afin de produire des résumés ou de vérifier si un jeu de données est équilibré (i.e. avec le même nombre d'observations par traitement).
On peut utiliser le paquet tidyr pour manipuler la structure d'un jeu de données en préservant toutes les informations d'origine à l'aide des fonctions suivantes:
- 1. “rassembler” les données (gather data en anglais; large –> long)
- 2. “disperser” les données (spread data en anglais; long –> large)
Supposons que vous envoyez votre assistant de terrain pour faire la collecte de données des dimensions de plusieurs arbres sur un site de recherche, soit le diamètre à la hauteur de la poitrine (DHP) et la hauteur. Il/elle revient avec un jeu de donnés en format “dégât” (format large):
- |
> large <- data.frame(Species = c("Chêne", "Orme", "Frêne"), DHP = c(12, 20, 13), Haut = c(56, 85, 55)) > large Species DHP Haut 1 Chêne 12 56 2 Orme 20 85 3 Frêne 13 55
gather(): Rassembler les données en longueur
- |
?gather
La plupart des paquets dans le “Hadleyverse” requièrent un format long, c'est-à-dire où chaque rangée est une observation unique, et chaque colonne est une variable. Utilisons la fonction gather() pour “rassembler” nos données en longueur. gather() prend plusieurs colonnes et les empile dans deux colonnes: une colonne spécifiant la variable mesurée, et l'autre spécifiant la mesure associée.
La fonction gather() prend au moins 3 arguments:
- data: le jeu de données (e.g. “large”)
- key: le nom de la nouvelle colonne spécifiant la variable mesurée (e.g. “dimension”)
- value: le nom de la nouvelle colonne spécifiant la mesure associée (e.g. “cm”)
- …: les colonnes qu'on veut empiler dans le jeu de données (e.g. “DBH”, “Haut”)
Voici une démonstration avec le jeu de données large utilisé précédemment, contenant les dimensions d'arbres:
- |
# Rassembler les colonnes en rangées d'observations uniques > long <- gather(large, dimension, cm, DHP, Haut) > long Species dimension cm 1 Chêne DHP 12 2 Orme DHP 20 3 Frêne DHP 13 4 Chêne Haut 56 5 Orme Haut 85 6 Frêne Haut 55
Essayons également avec le jeu de données CO2. On peut “rassembler” les colonnes contant la concentration de CO2 (conc) et le CO2 absorbé (uptake) :
spread(): Disperser les données en largeur
- |
?spread
La fonction spread() est l'inverse de gather(): elle nous permet de transformer nos données en format large, en utilisant la même syntaxe. spread() prend 3 arguments principaux:
- data: le jeu de données (e.g. “long”)
- key: nom de la colonne contenant les noms des variables (e.g. “dimension”)
- value: nom de la colonne contenant les mesures associées aux variables (e.g. “cm”)
- |
> large2 <- spread(long, dimension, cm) > large2 Species DHP Haut 1 Chêne 12 56 2 Frêne 13 55 3 Orme 20 85
separate(): séparer une colonne en deux (ou plusieurs) colonnes
Il arrive parfois qu'un jeu de données contient plusieurs variables réunies dans une même colonne. Dans ces situations, la fonction separate() sépare la colonne pour que chacune des variables ait sa propre colonne.
La fonction separate() prend 4 arguments, et sépare une colonne à partir d'une chaîne de caractères:
- data: le jeu de données (e.g. “long”)
- col: nom de la colonne que nous voulons séparer
- into: nom des nouvelles colonnes crées par la séparation
- sep: le caractère ou la chaîne de caractères identifiant où séparer le contenu (e.g. ” “)
À titre d'exemple, créons un jeu de données fictif sur les poissons et le zooplancton qui est mal organisé:
- |
set.seed(8) degat <- data.frame(id = 1:4, trt = sample(rep(c('controle', 'culture'), each = 2)), zooplancton.T1 = runif(4), poisson.T1 = runif(4), zooplancton.T2 = runif(4), poisson.T2 = runif(4))
Dans ce cas, on commence par “rassembler” ce jeu de données en largeur en format long.
Petit rappel rapide: ici, nous utilisons -id et -trt, qui est interprété par la fonction comme étant TOUTES les colonnes sauf id et trt. Nous pourrions également énumérer chaque colonne comme ceci: zooplancton.T1, poisson.T1, zooplancton.T2, poisson.T2.
Le défi principal de separate() est de formuler une expression qui identifie le caractère sur lequel séparer la colonne. Dans notre exemple, nous voulons séparer le contenu de la colonne “taxa” au point (.). Il faut donc utiliser un petit truc provenant des outils d'expressions régulières pour travailler avec les chaînes de caractères (strings en anglais):
- |
> degat.long.sep <- separate(degat.long, taxa, into = c("especes", "temps"), sep = "\\.") > head(degat.long.sep) id trt especes temps count 1 1 controle zooplancton T1 0.3215092 2 2 controle zooplancton T1 0.7189275 3 3 culture zooplancton T1 0.2908734 4 4 culture zooplancton T1 0.9322698 5 1 controle poisson T1 0.7691470 6 2 controle poisson T1 0.6444911
Ici, notons que la syntaxe \\. est requise parce qu'un point seul (.) est une carte frime (un joker) pour plusieurs fonctions dans le langage R. Les \\ servent donc à indiquer qu'on fait référence au caractère ”.“.
Récapitulatif: tidyr
tidyr est un paquet qui réorganise la structure de jeux de données.
- Convertir de format large en format long à l'aide de
gather() - Convertir de format long en format large à l'aide de
spread() - Séparer et regrouper des colonnes à l'aide de
separate()et de son inverse,unite()
Voici un aide-mémoire (en anglais) pour faciliter la manipulation de jeux de données avec tidyr et dplyr: https://www.rstudio.com/wp-content/uploads/2015/02/data-wrangling-cheatsheet.pdf
DÉFI: Utiliser tidyr pour convertir le format long en format large
Réorganisez le jeu de données airquality en format long (en rassemblant toutes les colonnes sauf “Month” et “Day”). Ensuite, dispersez-le en format large pour retrouver le format original de airquality:
Utiliser "dplyr" pour manipuler les données
Introduction à dplyr
Souvent, on doit faire appel à une gamme d'outils complexes pour manipuler nos jeux de données. La mission de dplyr est de simplifier nos tâches de manipulation en regroupant toutes les opérations communes sous un même toit. Le résultat est une collection de fonctions ayant une syntaxe simple qu'on peut exécuter à l'aide de “verbes” (ou fonctions) intuitifs.
En plus d'être facile à utiliser, le paquet dplyr est exceptionnel pour les raisons suivantes:
- il peut traiter des grands jeux de données rapidement (écrit en langage
Cpp) - sa fonctionnalité est bien intégrée dans RStudio et dans les autres paquets du Hadleyverse
- il peut établir des connections à des bases de données SQL, et traduire votre code en requête SQL
- c'est le Batman des paquets
R(maîtrise la peur des données et adopte des technologies avancées)
Certaines fonctions de base dans R ressemblent à des fonctions dans dplyr, incluant: split(), subset(), apply(), sapply(), lapply(), tapply() and aggregate().
Commençons par installer et charger le paquet dplyr:
- |
if(!require(dplyr)){install.packages("dplyr")} library(dplyr)
Au coeur du paquet dplyr, on retrouve des “verbes” essentiels qui nous permettent d'accomplir la manipulation de données. Voici 4 verbes qui exécutent les opérations les plus communes:
select(): sélectionne des colonnes dans un jeu de donnéesfilter(): filtre des rangées suivant les critères spécifiésarrange(): trie les données d'une colonne en ordre croissant ou décroissantmutate(): crée une nouvelle colonne de données (ou transforme une colonne existante)
Dans les exemples et les défis qui suivent, nous utiliserons les jeux de données airquality et ChickWeight, qui sont inclus dans R:
select(): Sélection de colonnes
La syntaxe de la fonction select() est: select(données, colonne1, colonne2, …). La majorité de fonctions dplyr suivent cette même syntaxe. select() prend alors au moins 2 arguments:
- data: le jeu de données
- …: noms ou positions de colonnes, ou expressions complexes (séparés par des virgules pour désigner les colonnes que l'on veut sélectionner
Par exemple:
- |
select(données, colonne1, colonne2) # sélectionne colonne1 et colonne2 select(données, c(2:4,6) # sélectionne les colonnes 2 à 4, plus la 6ème colonne select(données, -colonne1) # sélectionne toutes les colonnes sauf la 1ère select(données, start_with(x.)) # sélectionne les colonnes ayant un nom qui commence par "x."
Voici d'autres arguments utiles pour select():
À titre d'exemple, supposons qu'on s'intéresse à la variation de la variable Ozone avec le temps. À l'aide de la fonction select(), on peut retenir uniquement les colonnes requises pour nos analyses :
- |
> ozone <- select(airquality, Ozone, Month, Day) > head(ozone) Ozone Month Day 1 41 5 1 2 36 5 2 3 12 5 3 4 18 5 4 5 NA 5 5 6 28 5 6
filter(): Sélection de rangées
Souvent, on s'intéresse seulement à un sous-ensemble de notre jeu de données. Pour extraire un sous-ensemble de rangées selon une condition, on peut utiliser la fonction filter() avec la syntaxe suivante: filter(dataframe, proposition logique 1, proposition logique 2, …).
On se rappelle que la réponse à une proposition logique est VRAI ou FAUX (TRUE or FALSE). La fonction filter() retient uniquement les rangées pour lesquelles la réponse à la proposition logique est VRAI. On peut aussi utiliser cette fonction sur des chaînes de caractères (strings) et des facteurs (factors).
Voici un petit rappel du fonctionnement des propositions logiques dans R:

Par exemple, supposons qu'on s'intéresse aux périodes de canicules du mois d'août dans le jeu de données airquality :
- |
> aout <- filter(airquality, Month == 8, Temp >= 90) > head(aout) Ozone Solar.R Wind Temp Month Day 1 89 229 10.3 90 8 8 2 110 207 8.0 90 8 9 3 NA 222 8.6 92 8 10 4 76 203 9.7 97 8 28 5 118 225 2.3 94 8 29 6 84 237 6.3 96 8 30
arrange(): Triage de rangées
Parfois, on travaille avec des jeux de données qui doivent être analysés et/ou visualisés dans un ordre en particulier (e.g. série temporelle).
La fonction arrange() permet de trier les rangées d'un jeu de données selon une ou plusieurs colonnes, suivant cette syntaxe: arrange(données, variable1, variable2, …).
Par défaut, les rangées sont réordonnées en ordre croissant (alphabétiquement ou numériquement). On peut également réordonner les rangées en ordre décroissant en enveloppant la variable d'intérêt par la fonction desc() au sein de la fonction arrange(), comme ceci: arrange(données, variable1, desc(variable2), …).
Commençons par créer une version désordonnée de airquality:
- |
> air_degat <- sample_frac(airquality, 1) > head(air_degat) Ozone Solar.R Wind Temp Month Day 21 1 8 9.7 59 5 21 42 NA 259 10.9 93 6 11 151 14 191 14.3 75 9 28 108 22 71 10.3 77 8 16 8 19 99 13.8 59 5 8 104 44 192 11.5 86 8 12
Maintenant, voici comment réarranger le jeu de données en ordre chronologique, soit en ordre croissant de Month et ensuite en ordre croissant de Day :
- |
> air_chron <- arrange(air_degat, Month, Day) > head(air_chron) Ozone Solar.R Wind Temp Month Day 1 41 190 7.4 67 5 1 2 36 118 8.0 72 5 2 3 12 149 12.6 74 5 3 4 18 313 11.5 62 5 4 5 NA NA 14.3 56 5 5 6 28 NA 14.9 66 5 6
Quelle est la différence quand on change l'ordre des colonnes ciblées par la fonction?
- |
arrange(air_mess, Day, Month)
mutate(): Créer des nouvelles colonnes
Avant même de passer au triage ou à l'extraction d'un sous-ensemble de données, il faut souvent générer des nouvelles variables ou transformer des variables existantes. On peut utiliser la fonction mutate() pour créer et transformer des variables.
La fonction mutate() suit la syntaxe suivante: mutate(données, newVar1 = expression1, newVar2 = expression2, …).
Utilisons mutate() pour créer une nouvelle colonne. Par exemple, la température est en degrés Fahrenheit dans le jeu de données airquality, et nous voulons transformer la variable Temp en degrés Celsius:
- |
> airquality_C <- mutate(airquality, Temp_C = (Temp-32)*(5/9)) > head(airquality_C) Ozone Solar.R Wind Temp Month Day Temp_C 1 41 190 7.4 67 5 1 19.44444 2 36 118 8.0 72 5 2 22.22222 3 12 149 12.6 74 5 3 23.33333 4 18 313 11.5 62 5 4 16.66667 5 NA NA 14.3 56 5 5 13.33333 6 28 NA 14.9 66 5 6 18.88889
Notez que dans notre exemple, la syntaxe est très simple. Cependant, à l'intérieur d'un seul appel de la fonction mutate() on peut:
- remplacer le contenu de colonnes existantes (en conservant le même nom)
- créer plusieurs nouvelles colonnes
- créer des nouvelles colonnes calculées à partir de variables créés en amont
dplyr et magrittr, un don du ciel
Le paquet magrittr introduit un nouvel outil commun dans plusieurs langages informatiques : le pipe. Le “pipe” (un tuyau en français) permet de relier nos fonctions en redirigeant la sortie (output) d'une fonction en amont vers l'entrée (input) d'une fonction en aval. Le “pipe” fournit par le paquet magrittr s'écrit come ceci : %>%. Lorsqu'on l'utilise en combo avec dplyr, le “pipe” de magrittr nous permet d'accéder à la flexibilité et à toutes les fonctionnalités intéressantes du paquet. Pour vous familiariser avec ce type de flux de travail, nous utiliserons dplyr avec magrittr pour le restant de l'atelier.
Commençons par installer et charger le paquet :
- |
if(!require(magrittr)){install.packages("magrittr")} library(magrittr)
L'utilisation de magrittr est très intuitif. Nous allons démontrer son fonctionnement en combinant les exemples ci-haut. Supposons qu'on veut créer un sous-ensemble de airquality pour le mois de juin, et ensuite convertir la variable de la température en degrés Celsius. Si on procède sans le “pipe”, soit une étape à la fois, notre code lirait comme suit :
- |
juin_C <- mutate(filter(airquality, Month == 6), Temp_C = (Temp-32)*(5/9))
Ce code est plus difficile à lire parce que l'ordre des opérations exécutées commence au centre et se lit vers l'extérieur, jusqu'à la fonction enveloppant le tout. Plus on ajoute des opérations, plus le code devient illisible. Au lieu d'envelopper tous les fonctions, on peut écrire les opérations en ordre d'exécutions et les relier à l'aide du “pipe” %>% :
- |
juin_C <- airquality %>% filter(Month == 6) %>% mutate(Temp_C = (Temp-32)*(5/9))
Notez qu'au lieu de spécifier le jeu de données au sein de chaque fonction, on inscrit le jeu de données avant les opérations et on le “pipe” jusqu'à la prochaine fonction à l'aide de %>%.
En utilisant le “pipe”, le code est moins redondant. De plus, il se lit et s'écrit dans le même ordre que l'exécution des opérations, ce qui facilite et accélère la traduction de nos pensées en code, ainsi que la lecture et la compréhension du code écrit par nos collègues. Lorsque nos opérations de manipulation de données deviennent plus complexes et requièrent plusieurs étapes, on s'aperçoit rapidement que magrittr offre une approche puissante et élégante pour la rédaction de notre code dplyr.
BONUS : Dans RStudio, on peut insérer le “pipe” rapidement avec la combinaison des touches suivantes: Ctrl (or Cmd sur Mac) +Shift+M.
Regroupement d'opérations et sommaires avec "dplyr"
Les verbes dplyr que nous avons utilisés dans cet atelier sont utiles individuellement, mais deviennent particulièrement puissants quand ils sont reliés par le “pipe” (%>%), et lorsqu'on les applique sur des sous-ensembles de données. Les fonctions dplyr suivantes nous permettent de séparer nos jeu de données en groupes distincts sur lesquels on peut exécuter des opérations individuelles, comme des fonctions d'aggrégation et de sommaire:
group_by(): regrouper le jeu de données par un facteur pour les opérations en aval (commesummarise)summarise(): créer un sommaire de variables au sein de groupes distincts dans un jeu de données en utilisant des fonctions d'aggrégation (e.g.min(),max(),mean(), etc…)
Ces deux verbes fournissent la structure requise pour la stratégie Séparer-Appliquer-Combiner (“Split-Apply-Combine”) originalement introduite dans le paquet plyr, l'ancêtre de dplyr.
Utilisons ces deux fonctions pour générer un sommaire du jeu de données airquality qui montre la température moyenne et l'écart type pour chaque mois:
- |
> mois_moy <- airquality %>% group_by(Month) %>% summarise(mean_temp = mean(Temp), sd_temp = sd(Temp)) mois_moy Source: local data frame [5 x 3] Month mean_temp sd_temp (int) (dbl) (dbl) 1 5 65.54839 6.854870 2 6 79.10000 6.598589 3 7 83.90323 4.315513 4 8 83.96774 6.585256 5 9 76.90000 8.355671
DÉFI: dplyr et magrittr
En utilisant le jeu de données ChickWeight, créez un tableau sommaire dans lequel on retrouve la différence de masse entre le maximum et le minimum de la masse enregistré pour chaque poussin dans l'étude. Utilisez les verbes dplyr et le “pipe” %>%.
Indice pour les ninjas
Notez qu'on peut regrouper un jeu de données par plus qu'un facteur, en suivant la syntaxe suivante :
group_by(groupe1, groupe2, …)
Au sein de “group_by()”, on peut regrouper par plusieurs variables pour créer un “oignon” de sous-groupes. Chaque fois qu'on utilise la fonction summarise(), elle élimine une couche de l'oignon en commençant par la couche extérieure, donc la dernière variable dans group_by() qui s'agit de groupe2 dans cet exemple. Ainsi, dans l'exemple ci-haut, si on utilise la fonction summarise() une seule fois en aval:
group_by(groupe1, groupe2) %>% summarise(x = mean(y))
Le groupe2 est éliminé, et le jeu de données devient ensuite groupé par groupe1.
NINJA DÉFI: dplyr & magrittr
En utilisant le jeu de données ChickWeight, créez un tableau sommaire qui indique pour chaque Diet, la moyenne de la différence de masse entre la fin et le début de l'étude pour chaque poussin. Utilisez les verbes dplyr et le pipe %>%. (Indice : les fonctions first() et last() pourraient s'avérer utiles).
Fusionner des tableaux de données avec dplyr
En plus des fonctions que nous avons explorées aujourd'hui, dplyr offre d'autres fonctions forts utiles nous permettant de fusionner des tableau de données, avec une syntaxe relativement simple :
left_join()right_join()inner_join()anti_join()
Ces fonctions vont au-delà du matériel d'introduction dans cet atelier, mais ils offrent des fonctionnalités pouvant être très utiles pour des manipulations de données plus complexes.
Ressources supplémentaires
- r_atelier2.txt
- Last modified: 2021/10/13 23:49
- by lsherin